CRISPR文库服务
CRISPR文库是基于CRISPR/Cas9技术建立的高通量基因筛选方案,通过功能性筛选、富集以及深度测序分析挖掘与表型相关的基因或筛选药物新靶点。筛选的范围可以是全基因组,也可以是某个基因家族或者某个信号通路基因等。CRISPR/Cas9的多功能性、低噪声、高敲除效率和较小的脱靶效应,使得CRISPR文库筛选成为大规模基因功能筛选的首选平台。
CRISPR-iScreen™技术
源井生物基于自主研发的CRISPR-iScreen™技术,可提供CRISPR-KO、CRISPRa、CRISPRi三大定制文库从高通量sgRNA文库构建到病毒包装、细胞转染、药物筛选、高通量测序和数据分析等一站式服务,多种交付方式满足不同科研需求。

源井CRISPR-iScreen™ 技术优势
CRISPR-iScreen™是源井生物自主研发的一项创新技术,旨在实现高效的CRISPR筛选。该技术为科学家提供了一种高效、精准的工具,可应用于基因功能研究和药物靶点筛选。
自主研发高效感受态
使用自主研发的文库特制感受态细胞,更易捕获外源DNA,转化效率高且突变风险低,确保文库质粒扩增质量,覆盖度>99%,均一性<10
全流程优化的Cell Pool制备工艺
全流程优化的Cell Pool制备工艺,实现文库Cell Pool规模化和标准化生产,做到批间差异小、重复性好,Cell Pool文库覆盖度高达99%
多样化的筛选平台
源井生物提供一站式CRISPR体内+体外筛选服务,支持灵活配置多种实验压力条件(化合物处理、连续传代、病毒感染、流式等),并可选择多种富集策略。平台充分满足科研人员自主设计(DIY)功能筛选体系。
iScreenAnlys™文库分析平台
自主研发的交互式分析平台,界面友好,零门槛上手;同时支持:多种统计方法与可视化图表输出;个性化分析定制;一键生成可发表级别的图文结果。
CRISPR文库服务详情
CRISPR文库质粒
涵盖gRNA设计、引物芯片合成、载体构建、质粒提取及NGS验证(覆盖度>99%,均一性<10),更有40+现货文库质粒供您灵活选择,一站式助力高效CRISPR筛选!
CRISPR文库病毒 & Cell Pool
第三代高安全性病毒包装系统,病毒滴度≥1×10⁸ TU/ml;低MOI感染构建高质量Pool细胞,药筛一步到位。更有600+现货文库Cell Pool,筛选更灵活、实验更高效!
CRISPR文库体外筛选
多种压力施加与表型富集方式灵活组合,打造个性化功能筛选体系。源井生物多元表型筛选平台,精准应对各类科研需求,助力高效发现关键靶点!
体内筛选
CRISPR文库体内筛选能够在生物体中真实还原微环境与动态生理过程,精准解析基因功能的真实作用机制。源井生物已成功完成多项体内筛选项目,为科研创新提供有力技术支撑。
NGS测序
收集Baseline/NC/Sample样本,提取DNA进行NGS测序与gRNA差异分析,源井生物为您精准挖掘潜在靶点,助力科研突破!
CRISPR文库技术服务路线
准备阶段

gRNA设计

Oligo Pool合成

质粒文库构建

病毒包装
筛选阶段

Cell Pool制备 (病毒库感染细胞)

细胞筛选 (施加筛选压力进行正向或负向筛选)
分析阶段

PCR扩增

扩增子测序

数据分析 找出潜在靶点
准备阶段
gRNA设计
Oligo Pool合成
质粒文库构建
病毒包装
筛选阶段
Cell Pool制备 (病毒库感染细胞)
细胞筛选 (施加筛选压力进行正向或负向筛选)
分析阶段
PCR扩增
扩增子测序
数据分析 找出潜在靶点
应用案例
肿瘤功能基因筛选
阳性筛选Vemurafenib的耐药基因
Vemurafenib是突变的蛋白激酶BRAF的抑制剂,被批准用于晚期黑色素瘤的治疗,对存在V600E BRAF突变的黑色素瘤有治疗效果。Shalem等[1]设计了靶向全基因组18080个基因、包含64751条sgRNA的GeCKO敲除文库,而后构建慢病毒载体将GeCKO文库转染进携带V600E BRAF突变的黑色素瘤细胞A375中,通过加入Vemurafenib抑制A375细胞生长来富集少数具有Vemurafenib药物抗性的细胞。对这部分细胞的sgRNA序列进行分析,筛选出nf1、med12、nf2、cul3、tada2b和tada1等敲除后能使细胞对Vemurafenib产生抗性的基因,提出了新的Vemurafenib肿瘤耐药机制假说。


阴性筛选合成致死的基因组合
合成致死指当两个非致死性突变基因单独发生时不会导致细胞死亡,而同时发生时会引起细胞死亡的现象,是目前抗肿瘤药物领域新的研究方向之一。因为肿瘤细胞往往带有许多点突变,如何特异性地杀伤突变率高的肿瘤细胞而不影响正常细胞是抗肿瘤药物研发的一大追求。Shen等[2]从合成致死这一思路出发,设计了双重gRNA文库筛选出合成致死相互作用网络。与一般的sgRNA文库不同,双重gRNA文库中每个载体包含两条gRNA,一条靶向肿瘤中常见的突变肿瘤抑制基因,另一条靶向可以被抗癌药物扰乱的基因。他们运用该系统对3种实验癌细胞系(人宫颈癌Hela、肺癌A549和胚胎肾细胞癌293T)中的73个基因进行了筛选,共约150,000种基因组合,通过检测不同时间点gRNA丰度变化,进一步分析筛选出120种合成致死相互作用关系,为癌症新药的开发提供了新靶点。
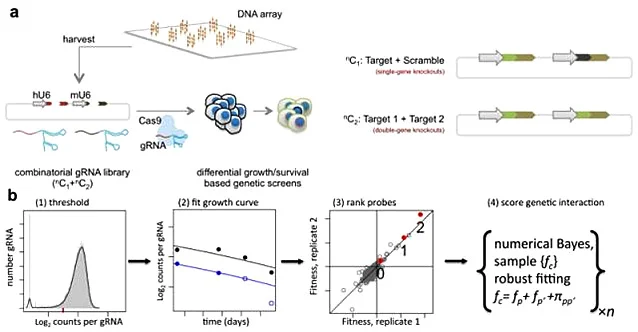
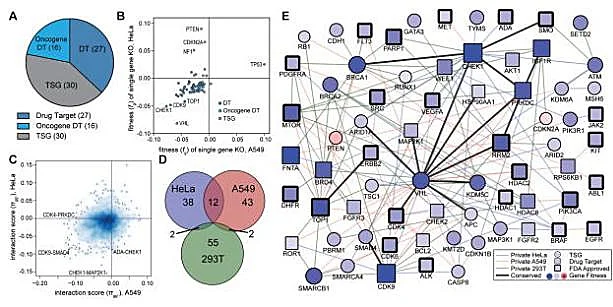
病毒感染基因筛选
筛选HIV治疗靶点
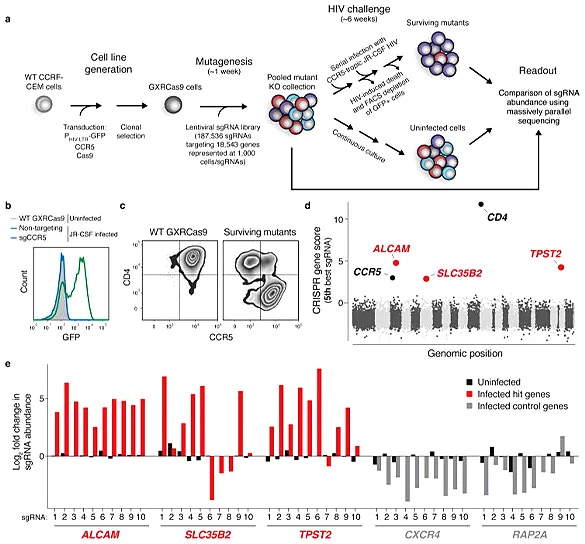
HIV病毒感染引发的艾滋病(AIDS)严重威胁着人类的生命健康,阐明HIV突破宿主细胞防御系统的分子机制、开发HIV治疗新靶点具有重大意义。Park等[3]在CCR5-hygR和HIV-1 LTR-GFP稳定表达的CCRF-CEM细胞上感染Cas9病毒,筛选出一株HIV感染前高表达CCR5、低表达EGFP但感染后高表达EGFP的克隆株(GXRCas9细胞)用作筛选HIV感染靶点的工具细胞。具体是使用包含187536条sgRNA (靶向18543个基因)的慢病毒文库感染GXRCas9细胞,再用HIV病毒株JR-CSF感染这些缺失不同受体的T细胞,随后流式分选出GFP阴性和阳性的T细胞,并对GFP阴性细胞群与未感染HIV病毒细胞群进行测序,分析两群细胞sgRNA的丰度差异,最后筛选出5个sgRNA丰度变化最大的基因。其中CD4和CCR5是HIV感染T细胞的受体,TPST2和SLC35B2对CCR5进行修饰为HIV的结合提供便利,而编码白细胞粘附因子ALCAM的基因则与HIV在细胞间的传播有关。筛选出的5个基因被敲除后均不影响T细胞的存活,但能使T细胞抵抗HIV的感染,因此这5个基因可作为HIV治疗的潜在靶标,为预防和治疗艾滋病提供新的理念和途径。
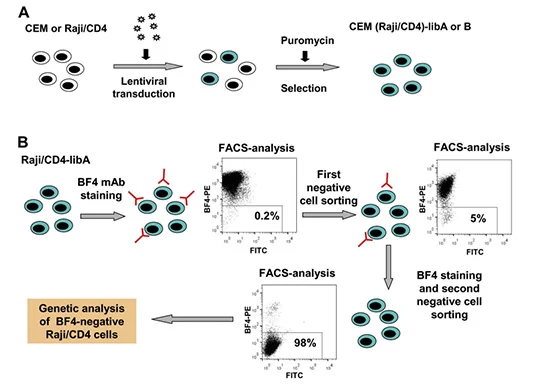
抗体靶标筛选
单克隆抗体特异性靶标抗原及其表位的识别
使用肽或纯化蛋白进行免疫实验验证抗体是相当简单的,但使用全细胞或其他复杂抗原进行免疫验证抗体则相对困难。如果Western blotting(蛋白印迹)和免疫沉淀中均未检测到抗体的反应性,则需要应用多种基因操作层面的技术来确定mAb的抗原特异性。BF4是一种能与未感染的淋巴细胞、中性粒细胞及HTLV-1感染细胞表面的病毒生物膜结合的抗体。Zotova等[4]以MT2细胞(人T细胞嗜淋巴病毒Ⅰ型HTLV-1慢性感染T细胞)作为免疫原触发小鼠免疫获得了一株HTLV-1生物膜特异性单克隆抗体BF4。他们基于通过向BF4阳性细胞中转导CRSIPR knockout 文库以筛选出BF4抗原被敲除的细胞的思路,在CEM T和Raji/CD4 B细胞上转导GeCKO文库,将不与BF4结合的细胞分选出来。经过两轮的重复分选后,阴性细胞比例达到了99%以上。研究人员对这部分细胞进行测序分析,发现约80%的sgRNA靶向了CD82。经验证确定BF4是CD82的特异性抗体。
FAQs
1. 做文库筛选客户需要提供什么?
1) 筛选目标与实验目的
- a) 明确筛选类型:正向筛选 / 负向筛选
- b) 希望研究的通路、表型、功能或靶点(如细胞增殖、抗药性、凋亡、迁移等)
2) 使用的细胞系信息
- a) 细胞名称、来源、是否贴壁、生长特性
- b) 是否已有Cas9表达
- c) 转导效率、抗生素敏感性(如puromycin浓度范围)
3) 文库选择偏好(如有)
- a) 全基因组文库 / Pathway文库 / 自定义文库
- b) 是否已有目标基因清单,或需源井生物协助设计
4) 筛选策略与处理方案
- a) 筛选方式(如化合物处理、传代培养、流式分选、体内筛选)
- b) 筛选周期及富集时间点(如2周、4周等)
5) 是否需要下游测序与数据分析
- a) 源井生物为客户提供NGS测序和富集分析(如MAGeCK分析等)
6) 其他特殊要求(如有)
- a) 多轮筛选/重复实验设计
- b) 特定阳性/阴性对照设置
如客户尚未明确实验设计,源井生物可提供 一对一方案定制服务 ,协助优化筛选策略与文库选择。 立即咨询>>>
2. CRISPR文库筛选的原理是什么?
CRISPR文库筛选 是一种 大规模功能基因筛选技术 ,通过将包含数千至上万个不同sgRNA的文库导入细胞群体,实现对基因组范围内 每个基因的系统敲除、激活或抑制 。在特定选择压力下(如化合物筛选、流式分选等), 具有相应表型 的sgRNA会在筛选后富集或耗竭,通过NGS测序分析sgRNA的富集或耗竭情况,从而识别关键调控因子。
3. CRISPR文库筛选的步骤是什么?
1) 选择合适的细胞模型
- a) 要求可稳定表达 Cas9 或 dCas9(用于敲除/激活/干扰)
- b) 可满足病毒感染、具有一定的传代能力,并能承受筛选条件(如化合物、低氧等)
2) 文库构建与包装
- a) 选择合适的sgRNA文库(全基因组、通路特异、自定义)
- b) ○构建文库质粒,并包装为慢病毒,保证高质量、低偏差传递到细胞
3) 文库感染与建库
- a) ○低MOI感染目标细胞,保证每个细胞仅携带一个sgRNA
- b) 建立包含足够覆盖度的细胞库(一般要求 >300x /500x覆盖度)
4) 施加筛选压力
- a) 对感染文库的细胞施加特定条件(如化合物处理、传代、流式分选)
- b) 设定处理组与对照组,进行富集筛选
5) 基因组DNA提取与NGS测序
- a) 提取细胞基因组DNA,扩增sgRNA条码区
- b) 进行高通量测序,获得筛选前后sgRNA的丰度变化数据
6) 生物信息分析与候选基因筛选
- a) 使用MAGeCK、edgeR等分析工具识别富集/耗竭的sgRNA
- b) 得出候选关键基因,进一步验证其功能
4. 为什么选择源井生物的CRISPR文库筛选服务?
- 1) 多样化的表型分析平台
源井生物的多样化表型分析平台可支撑多种功能筛选体系所需,全面覆盖化合物/病毒处理、传代培养、细胞共培养、流式分选、细胞迁移和贴壁、体内筛选等筛选操作。 - 2) 成熟稳定的细胞生物学平台
源井生物的细胞生物学平台满足大规模细胞培养需求,技术人员具备丰富的细胞培养经验,支持在各类细胞体系上开展功能筛选。 - 3) 全流程精细化服务
源井生物具备完善的预实验摸索和质量管理体系,能够提供全流程功能筛选服务和CRISPR筛选一站式服务,轻松衔接CRISPR筛选各个流程节点,保证实验连续性和成功率。 - 4) 专业技术和项目管理团队指导
源井生物具备博士级别的专业技术团队,拥有丰富的功能筛选体系相关专业知识和操作经验,手把手指导功能筛选的设计和操作。