CRISPR文库|解锁乳腺癌靶点筛选新思路

CRISPR文库|解锁乳腺癌靶点筛选新思路

乳腺癌简介
乳腺癌是女性最常诊断出的恶性肿瘤,也是全球女性癌症死亡的第二大原因,其研究和治疗进展一直是医学界关注的焦点[1]。根据肿瘤组织中受体分子的表达状态,乳腺癌可以分为:Luminal A型、Luminal B型、HER-2过表达型和三阴性乳腺癌[2]。其中,三阴性乳腺癌不表达常规的激素受体和HER-2,这使得传统的内分泌治疗和HER-2靶向治疗在这类患者中效果有限[3]。因此,需要更加深入研究乳腺癌,开发新型的治疗手段,为乳腺癌患者提供了新的治疗选择和希望。
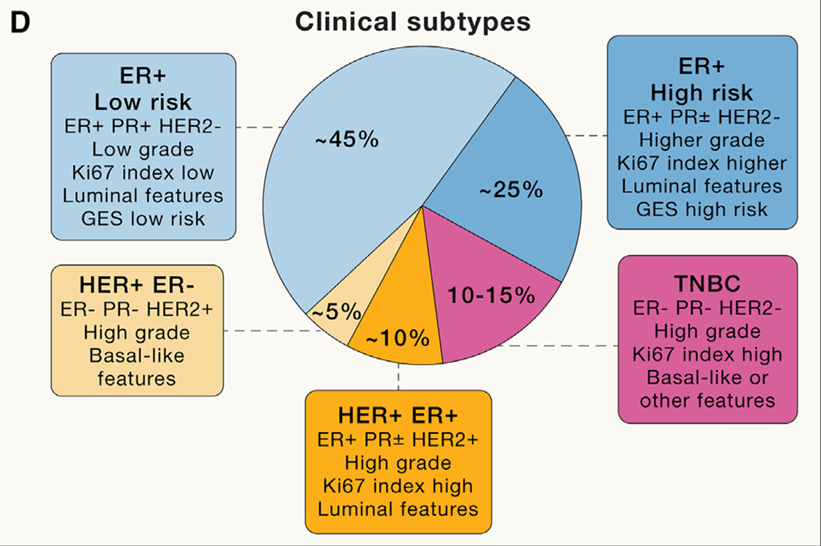
图1 乳腺癌亚型及占比[2]
随着基因编辑技术的飞速发展,CRISPR-Cas9系统以其卓越的精确性和灵活性,为癌症研究领域带来了革命性的变化。而CRISPR文库作为该技术的一种应用,通过大规模筛选基因功能,为乳腺癌的研究提供了强大的工具。跟随小源一起探索CRISPR文库筛选如何助力乳腺癌研究,我们可能会收获新的思路和见解,从而更加有效地应用这个强大的工具!
乳腺癌耐药新靶点的发现-NUMEN
BRCA1基因是与遗传性乳腺癌高度相关的肿瘤抑制基因,其功能丧失会导致DNA双链断裂修复缺陷,使得细胞对PARP抑制剂(PARPi)特别敏感,但是发现肿瘤细胞可能对PARPi耐药[4]。Chen Bohong等人[5]通过全基因组CRISPR文库筛选发现了NUMEN这一基因,它在BRCA1缺失的乳腺癌细胞中对PARPi的抗性至关重要。NUMEN通过其结构特异性的核酸酶活性,促进了非同源末端连接(NHEJ)依赖的DNA双链断裂修复。这项研究不仅揭示了NUMEN在DNA修复和基因组稳定性维持中的作用,还为开发针对PARPi耐药乳腺癌的新型治疗策略提供了可能的靶点。
文库类型:全基因组敲除CRISPR文库(GeCKOv2 库)
转导细胞:可诱导表达CAs9的BRCA1−/−基因敲除MDA-MB-231细胞
筛选思路:体外加药筛选
筛选方法:在含有多西环素(doxycycline)的条件下培养细胞,以诱导Cas9的表达,然后加入PARP抑制剂奥拉帕尼(olaparib)进行筛选。
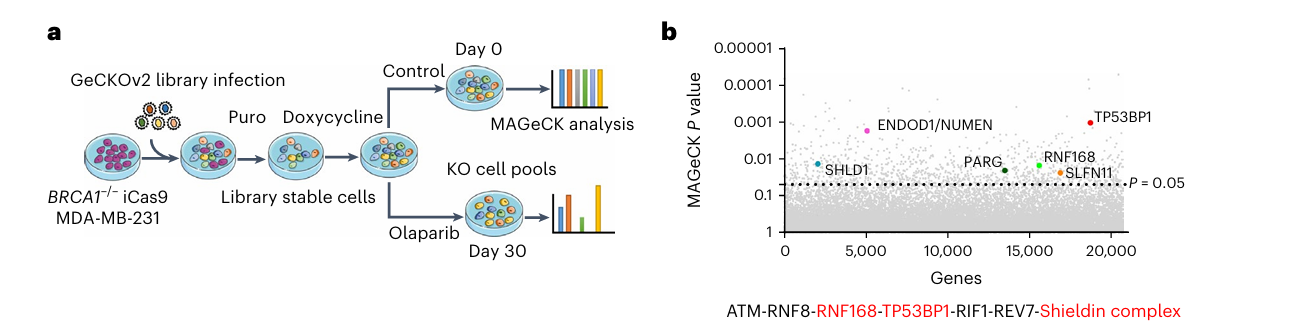
图2 全基因组CRISPR筛选流程[5]
三阴性乳腺癌靶点的发现-Cop1
三阴性乳腺癌(TNBC)是一种预后较差的癌症类型,对免疫检查点阻断(ICB)治疗的反应有限。而肿瘤微环境(TME)中的免疫细胞,特别是肿瘤相关巨噬细胞(TAMs),在促进肿瘤生长和转移中起着重要作用。Wang Xiaoqing等人[6]通过两轮CRISPR文库筛选,最终鉴定出E3泛素连接酶Cop1是一个重要的调节巨噬细胞浸润和抗PD-1反应的基因。Cop1的敲除可以减少肿瘤相关巨噬细胞的浸润,增强抗肿瘤免疫反应,并提高对ICB治疗的反应。通过抑制Cop1可以改善TNBC的癌症免疫治疗疗效,这为开发新的癌症治疗策略提供了潜在的靶点。
文库类型:基因敲除亚文库MusCK 1.0,基因敲除亚文库MusCK 2.0
靶向基因:MusCK 1.0:4500多个与肿瘤起始、进展和免疫调节相关基因;MusCK 2.0:在第一次文库筛选中确定的79个候选基因。
转导细胞:表达卵清蛋白(mOva)的4T1细胞
筛选思路:小鼠体内筛选,二次筛选。
筛选方法:为了模拟不同的免疫微环境,研究者使用了不同免疫状态下的小鼠,包括Foxn1nu/nu小鼠(免疫缺陷)、野生型小鼠(免疫能力正常)、接种了卵清蛋白(OVA)疫苗的小鼠。将转导后的4T1细胞注射到小鼠的乳房脂肪垫内,在肿瘤植入后的一定时间点,收集肿瘤组织。根据第一次筛选测序分析获得的79个候选基因再次构建sgRNA库,方法与第一次筛选类似并新增了接种OVA疫苗且接受PD-1阻断治疗的小鼠组。
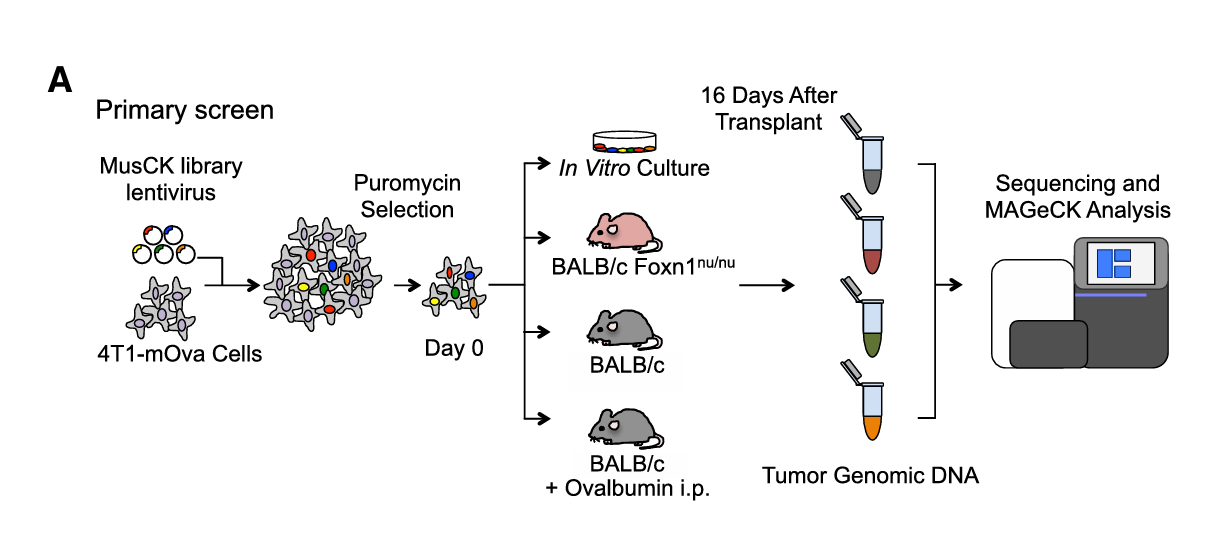
图3 第一次体内筛选流程[6]
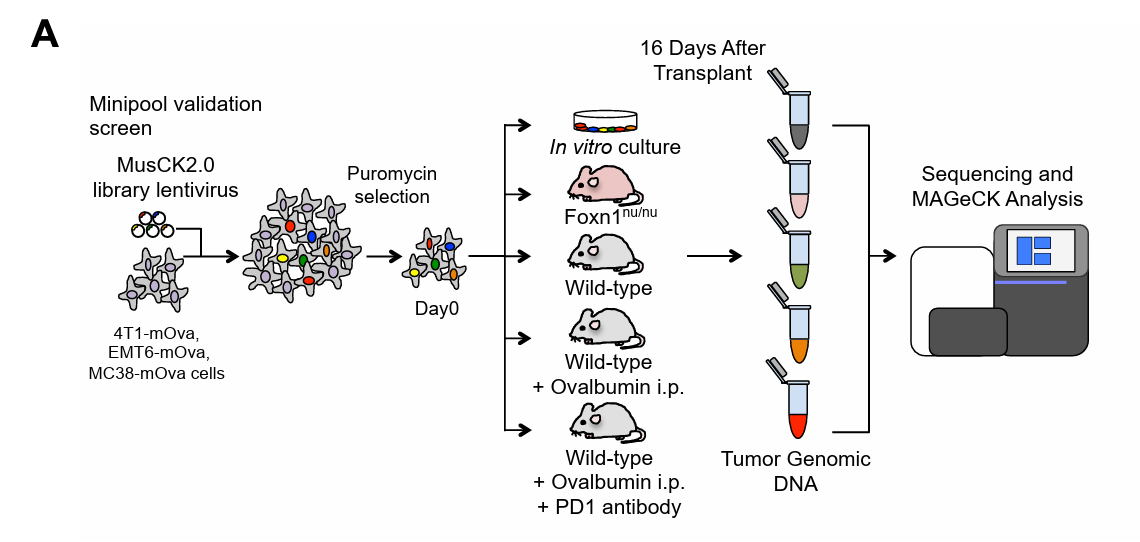
图4 第二次体内筛选流程[6]
乳腺癌风险基因识别新平台—CRISPR文库
Natasha K. Tuano等人[7]通过全基因组关联分析(GWAS)发现200多个与乳腺癌风险相关的基因,大多数候选致病突变位于非编码区,这些突变位点通过调控基因表达影响癌症表型。然而,精确定位靶基因并识别其表型是艰巨任务。为了克服这一挑战,他们进一步通过60个CRISPR文库筛选,最终鉴定出了20个高置信度的基因。这些基因通过促进乳腺细胞的增殖或调节DNA损伤响应来促进癌症的发生与进展。这一研究不仅展示了CRISPR文库筛选在准确识别风险位点基因靶点方面的能力,还证明了其高效性,为识别和增加乳腺癌风险相关的基因靶点提供了一个重要的平台。
文库类型:敲除文库、抑制文库以及激活文库。
靶向基因:由全基因组关联分析(GWAS)识别出的200多个与乳腺癌风险相关的位点基因。
转导细胞:六种不同乳腺细胞系,包括K5+/K19+、K5+/K19-、B80-T17、mesHMLE、B80-T5、HMLE
筛选思路:体外表型筛选,小鼠体内筛选,体外加药合成致死筛选
筛选方法:2D和3D细胞增殖筛选:在二维(2D)和三维(3D)培养条件下评估细胞的增殖能力,以模拟细胞在体内生长的情况。
体内筛选:将经过细胞注射到免疫缺陷的小鼠中,观察肿瘤形成情况。
DNA损伤响应筛选:使用PARP抑制剂奥拉帕尼(olaparib)处理细胞,筛选对DNA损伤响应有影响的基因。
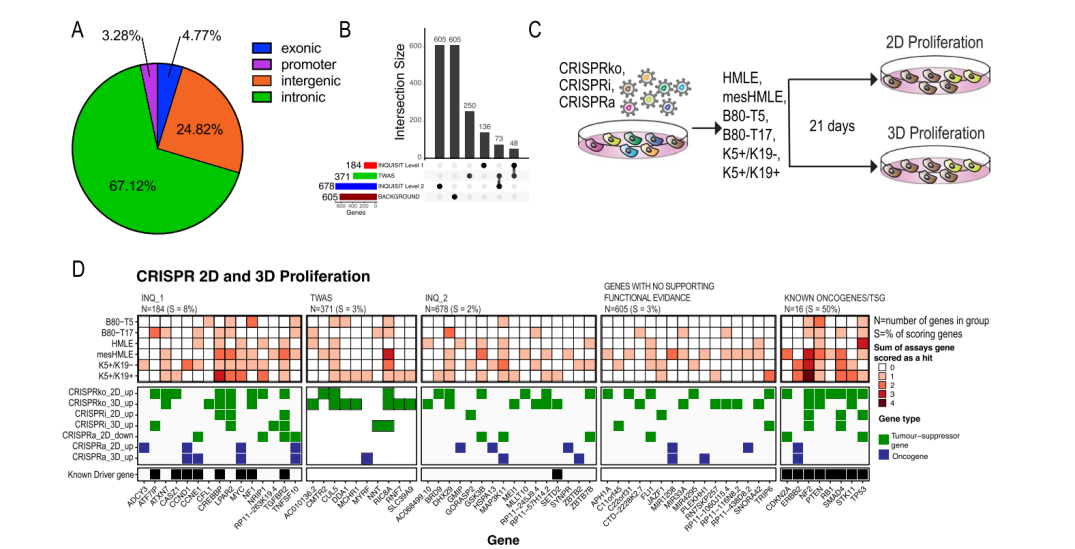
图5 CRISPR文库筛选确定了在2D、3D细胞培养模型中调节增殖的乳腺癌风险基因[7]
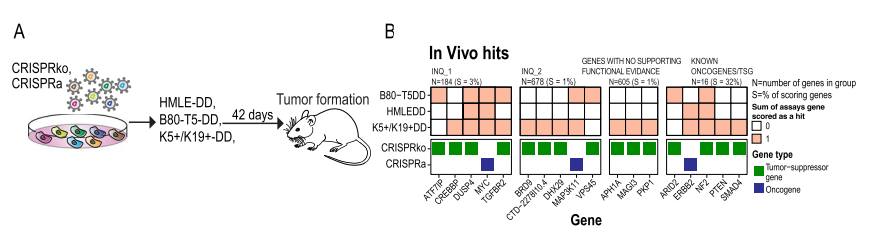
图6 CRISPR激活和抑制筛选确定调节小鼠肿瘤生长的乳腺癌风险基因[7]
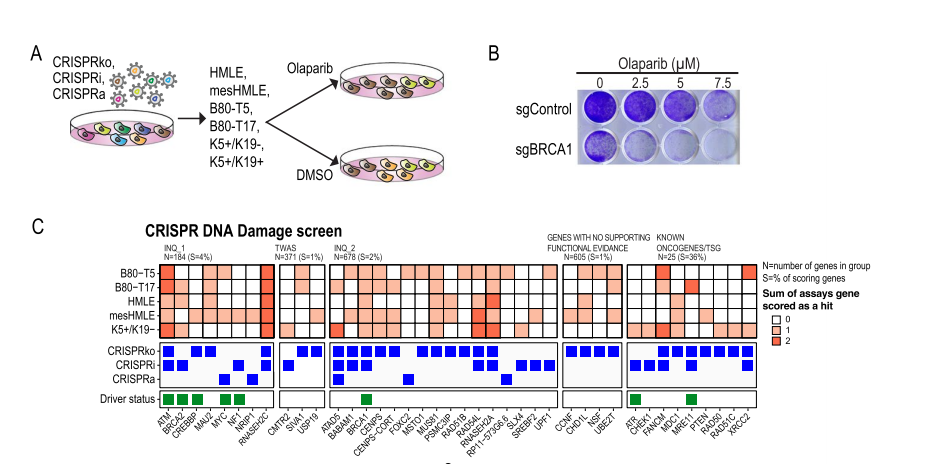
图7 Olaparib合成致死筛选识别调节DNA修复途径的乳腺癌风险基因[7]
小结
看完以上三个应用实例,我们不难发现,CRISPR文库在乳腺癌研究中具有多样性和灵活性。比如,在靶基因的选择上,不仅可以选择全基因组筛查,还可以选择其他方法筛选得到的大量基因。在细胞类型上,不仅可以使用野生型细胞,还可以选择基因编辑修饰的细胞以模拟特定基因突变的影响。在筛选方法上,可以是体外筛选,也可以是体内筛选,甚至可以采用体内和体外相互结合的筛选策略,以提高筛选的准确性和有效性。
总之,CRISPR文库技术为乳腺癌研究提供了个性化的、灵活的解决方案。根据具体的研究目标,可以对CRISPR文库进行个性化的调整和优化,从而筛选出置信度高的靶点,为乳腺癌的治疗开辟新的道路。
参考文献:
[1] https://www.who.int/zh/news-room/fact-sheets/detail/breast-cancer
[2] Nolan, E., Lindeman, G. J., & Visvader, J. E. (2023). Deciphering breast cancer: from biology to the clinic. Cell, 186(8), 1708–1728.
[3] Leon-Ferre, R. A., & Goetz, M. P. (2023). Advances in systemic therapies for triple negative breast cancer. BMJ (Clinical research ed.), 381, e071674.
[4] Geyer CE Jr, Garber JE, Gelber RD, et al. Overall survival in the OlympiA phase III trial of adjuvant olaparib in patients with germline pathogenic variants in BRCA1/2 and high-risk, early breast cancer. Ann Oncol. 2022;33(12):1250-1268.
[5] Chen, Bohong et al. Transmembrane nuclease NUMEN/ENDOD1 regulates DNA repair pathway choice at the nuclear periphery. Nature cell biology vol. 25,7 (2023): 1004-1016.
[6] Wang, Xiaoqing et al. “In vivo CRISPR screens identify the E3 ligase Cop1 as a modulator of macrophage infiltration and cancer immunotherapy target.” Cell vol. 184,21 (2021): 5357-5374.e22.
[7] Tuano NK, Beesley J, Manning M, et al. CRISPR screens identify gene targets at breast cancer risk loci. Genome Biol. 2023;24(1):59. Published 2023 Mar 29.










