更小规模满足更多场景:克隆优化让CRISPR文库筛选变得更紧凑
在CRISPR筛选实验中,sgRNA文库的均一性往往决定了结果的可靠性。然而,传统文库构建方式存在分布偏斜,导致部分sgRNA丰度过低甚至丢失。为避免假阴性,研究人员通常需要在极高细胞覆盖度(≥300×)下进行筛选。但这一要求对许多难以扩增的细胞类型(如iPSC衍生细胞、原代细胞)几乎不可行,严重限制了CRISPR筛选的应用范围。近期,Chow ED 团队在 Genome Biology 发表的研究(Heo SJ et al., 2024)提出了一种改进的sgRNA文库克隆策略——双向模板结合优化条件,构建出更紧凑、更均一的CRISPR文库(LGR library)。该方法显著降低了文库分布偏斜比(library skew ratio),使得在更低细胞覆盖度下仍能获得高质量的基因组筛选结果。

一、研究背景:从克隆偏差到筛选瓶颈
CRISPR筛选依赖sgRNA文库在细胞群体中均一分布。然而,常规文库在构建过程中存在克隆与扩增偏差,低丰度sgRNA往往在筛选过程中被淹没。为避免信息丢失,研究人员不得不提升细胞覆盖度,增加了实验成本和技术门槛。虽然部分研究尝试通过压缩文库设计(如双sgRNA系统)来减少规模,但会引入重组及统计复杂性。相比之下,Chow团队聚焦于文库克隆过程的优化,以更简洁的方式解决核心问题。
二、优化策略:实现更高质量的CRISPR文库
研究团队在逐步实验优化中总结出一套通用方案,使全基因组文库的sgRNA分布更为均一。
1. 模版合成:双向合成sgRNA文库Cell pool
以往研究中,研究人员常同时订购包含正义链和其反义链的sgRNA文库,但并未在方法中明确提及。此外,也并没有相应的数据支撑这种双向sgRNA文库模板的实际效用。而相较于单向sgRNA文库模板,Chow ED团队首次在研究中明确指出使用双向sgRNA文库模板进行克隆,并表明这种双向文库模板的克隆能够减少文库偏差和丢失(图1E、F)。
2. 模板克隆:适当减少扩增循环
Chow ED团队在比较了三种不同商业DNA聚合酶的合成质量后,择其最优进行后续文库的PCR克隆(图1A,B)。比较1次和15次PCR扩增循环的文库质量发现,额外的PCR扩增会导致微弱的文库偏差(补充图S4),以及非目的片段的过度扩增。而优化PCR循环次数以及模板浓度能够有效减少文库偏差以及非目的片段克隆。
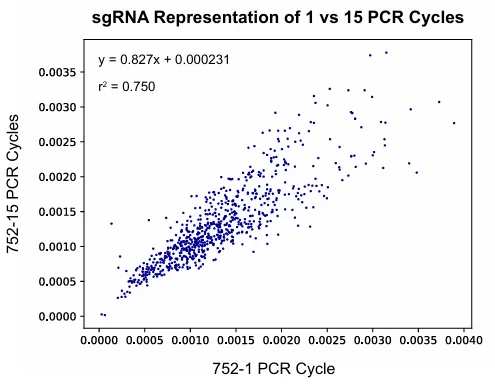
补充图S4. 额外的PCR循环导致微弱的sgRNA文库偏差
3. 片段插入:低温洗脱
在包含192个sgRNA的小文库中,37°的洗脱温度较70°的高温洗脱明显提升文库中sgRNA的均一性(图1A,B)。然而扩大到752个sgRNA文库时,37°的洗脱温度仍无法消除sgRNA本身Tm差异引起的文库偏差(图1C,D)。但当结合上述优化条件,并进一步的降低洗脱温度至4°时,即便是全基因组文库中sgRNA的 Tm分布与其丰度分布也不再具有显著差异(图1G)。
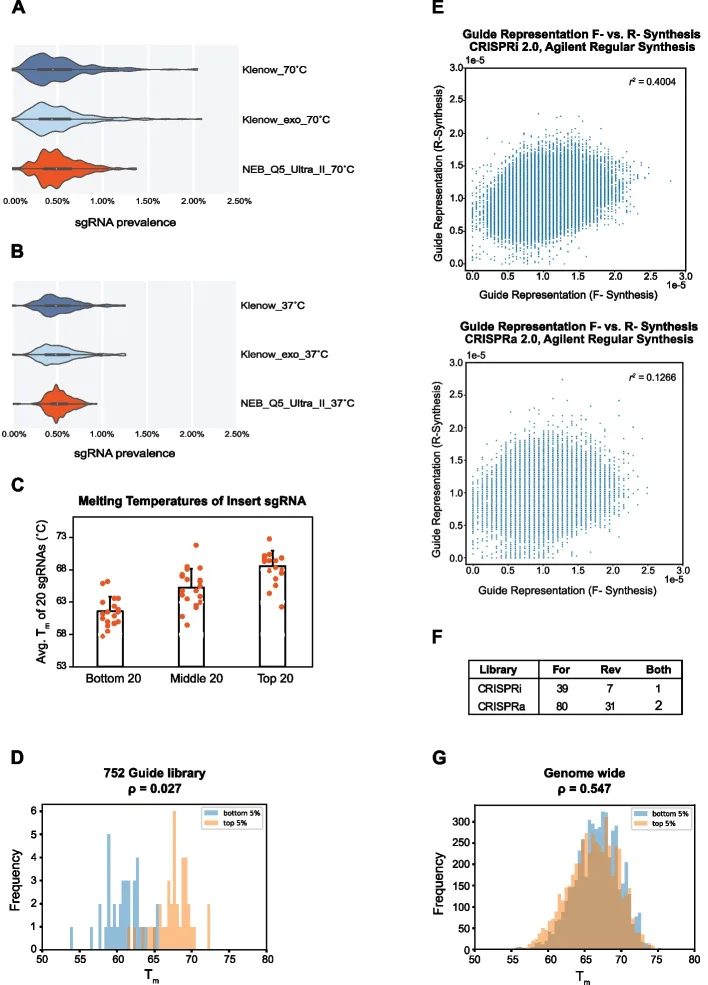
图1. 影响文库中sgRNA均匀性的因素
4. 文库克隆:单次整体克隆
传统的CRISPRi/a v2文库分为7个子文库独立克隆,每个子文库具有不同的文库偏斜,这使得文库整体偏斜受到方差较大的子库的影响。Chow ED团队结合优化条件直接一次性克隆整个全基因文库(LGR表示改进克隆方案形成的全基因组文库,Legacy表示传统克隆方案形成的全基因组文库),产生更加均一的sgRNA文库,且具有更少的sgRAN丢失(图2)。
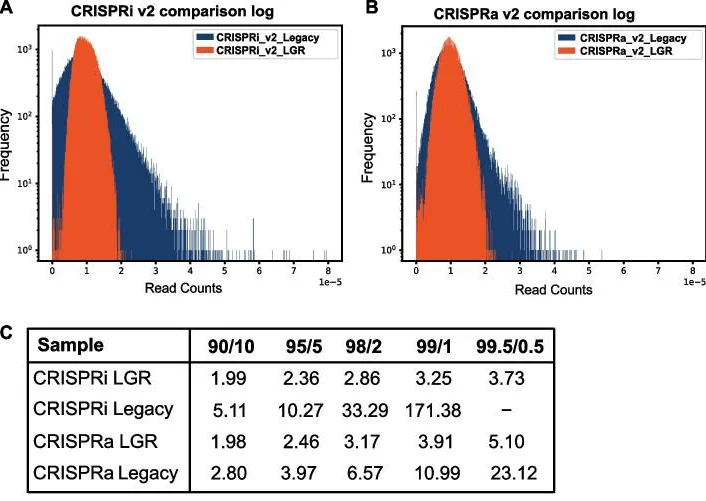
图2. 克隆优化的新方案改进了全基因组文库
三、效果展示:LGR文库具有更佳的筛选性能
1. LGR文库允许更低的细胞覆盖度
为了测试LGR文库能否在低细胞覆盖度下进行筛选,Chow ED团队通过使用LGR文库重现先前Horlbeck MA等人的全基因文库筛选研究(图3A)。通过比较必需基因的命中发现,LGR 1000×覆盖度下必需基因的命中数量与Horlbeck MA等人的原始研究相差不大,且有1366个必需基因的重叠(图3B,D)。更为重要的是, LGR 1000×覆盖度下AUC达0.937(图3C),这表明LGR文库具有高精度鉴定必需基因的能力。此外,LGR 100×覆盖度下也拥有AUC达0.949的精确度,且与LGR 1000×具有更多的命中重叠以及相似的命中基因表型评分(图3C、D和E)。
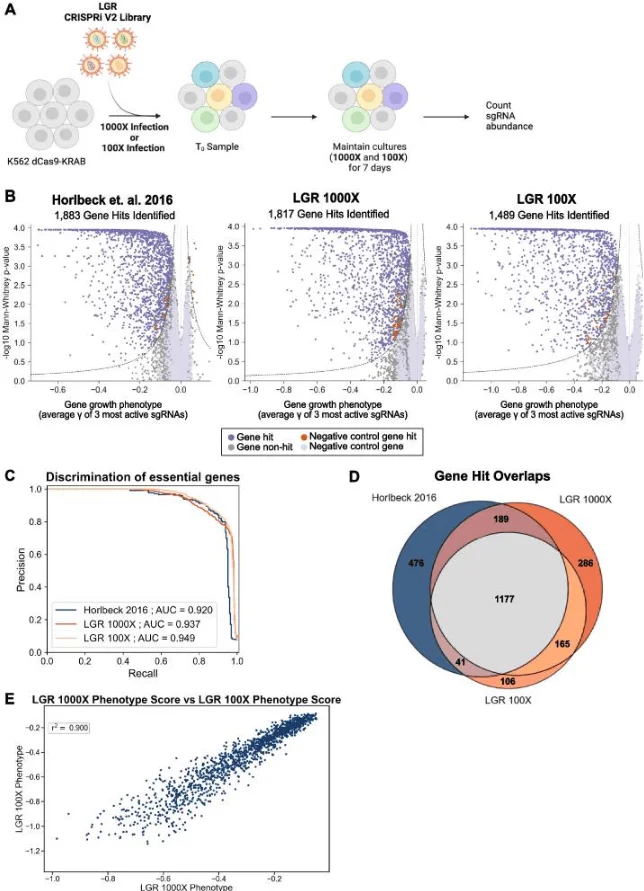
图3. 较低细胞覆盖度下优化后的LGR文库也有与现有文库相近的性能
在较低细胞覆盖度下进行筛选的一个主要挑战是sgRNA 丢失,LGR文库在200×、100×甚至50×细胞覆盖度下具有几乎一致的文库偏斜比和sgRNA丢失(图4),这表明理论上LGR文库可以在低至50×的细胞覆盖度下进行全基因组规模筛选。
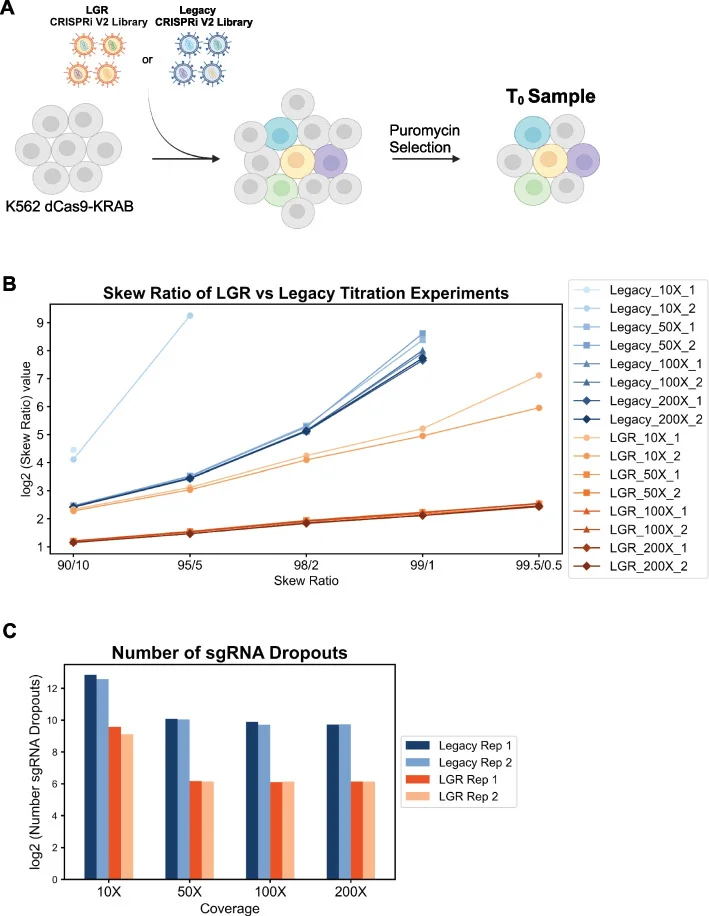
图4. LGR文库与传统文库转导后sgRNA均一性和丢失数比较
2. LGR文库产生更多候选基因命中
为了测试LGR文库是否能够在低覆盖度下实现高质量筛选,Chow ED团队在传统CRISPRi文库和LGR文库上进行达沙替尼(Dasatinib)药物生存筛选,以比较全面识别与达沙替尼耐药性和敏感性相关的基因的能力(图5A,B)。结果显示,LGR文库较传统文库具有更多必需基因命中,以及相近的药物相关基因命中(图5C,D)。并且随着FDR不断缩小,LGR文库具有更多候选基因命中(图5E)。
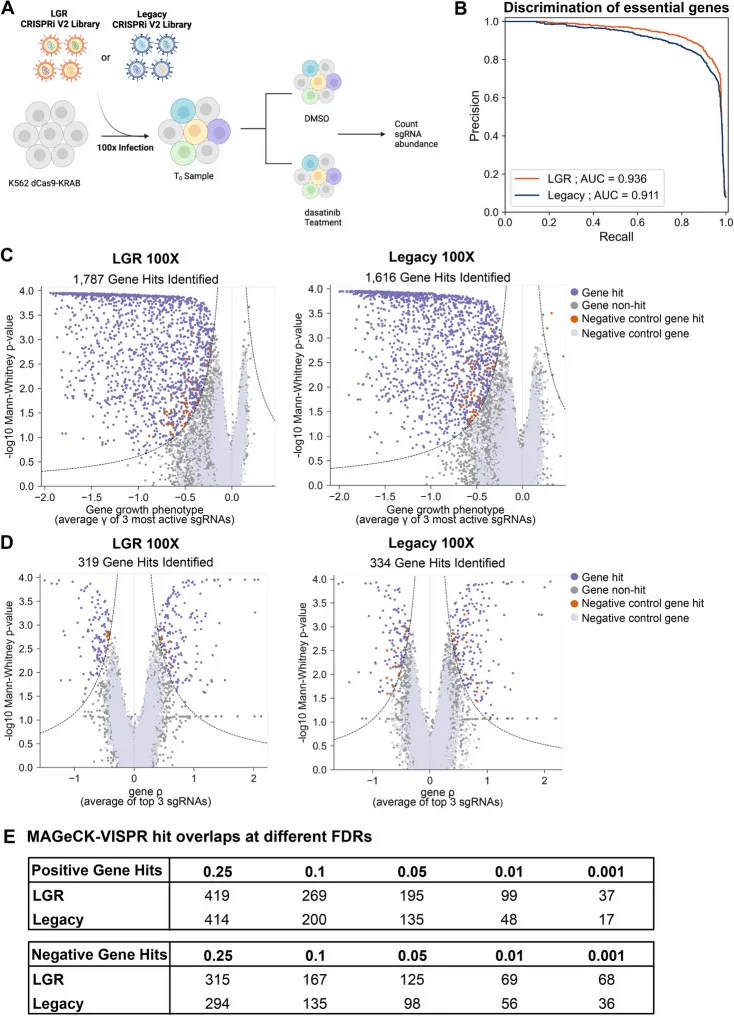
图5. LGR文库在100×覆盖度下产生更多候选基因命中
介质复合物(GO ID:0016592)和氧化磷酸化途径(KEGG ID:hsa00190)已被证明是与达沙替尼协同治疗的潜在药物靶点。而更为重要的是,LGR文库在介质复合物和氧化磷酸化途径相关基因中产生更多命中(图6),这进一步表明LGR文库在具有高精确的同时,能够带来更多筛选命中。
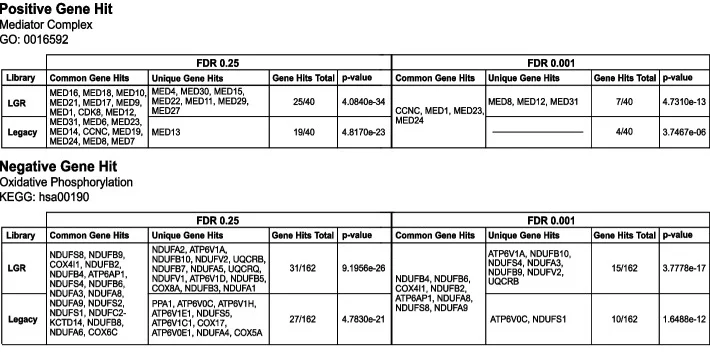
图6. LGR 和传统文库确定的介质复合物(上)和氧化磷酸化途径(下)的基因命中列表
四、技术优势与意义
综上所述,这种改进的克隆方案带来了显著的优势:
1. 细胞用量大幅减少
改进后的克隆方案使得sgRNA文库的均一性更高,这使得相同文库大小下,研究人员可以使用低至50×的细胞覆盖度来进行全基因组层面的筛选。而从另一个角度出发,研究人员可以在相同细胞量下进行更大规模的sgRAN筛选。
2. 筛选结果更可靠,假阴性/阳性减少
由于sgRNA分布更加均匀,减少了因sgRNA丢失或表达水平差异导致的假阴性或假阳性结果,这使得筛选结果更加准确可靠。同样的,研究人员可以在相同细胞量下进行更多次的生物学重复,这同样能够使得筛选结果更加准确可靠。
3. 拓展了CRISPR文库筛选的应用范围
一方面,这种改进的克隆方法使得在技术上具有挑战性的细胞或筛选模型式中进行基因组规模的CRISPR筛选成为可能,例如基于FACS的成像和单细胞测序方法的贴壁细胞、iPSC衍生细胞和原代细胞等模型。另一方面,这种简单的改进可以推广到多种CRISPR系统(如Cas9、Cas12、Cas13)及衍生技术(如碱基编辑、CRISPRoff等)。
如果你也在为传统CRISPR文库筛选投入庞大细胞量、耗费实验周期而苦恼,现在,源井生物为你带来“紧凑高效”的全新解决方案。借助我们CRISPR-iScreen™技术平台 + 源井体外/体内筛选服务,再加上零代码 iScreenAnlys™ 文库分析平台,你可以:
- · 直接调取源井Screening-ready Cell Pool (400+),省去构建等待阶段,实验即开即用;
- · 在体外模拟与体内真实环境中均可开展筛选,重现疾病微环境,精准锁定关键基因靶点;
- · 使用 iScreenAnlys™ 文库分析平台一键导入数据,无需编程基础即可自动进行质控、差异分析、通路富集与靶标排序,并生成期刊级火山图、热图等图表。
目前,源井文库体外筛选服务最低至 ¥15,000,最快 6 周完成。用更少的细胞量,更低的成本,更快的出图速度,让你在科研项目中抢占“时间 + 精度”的领先优势。
联系我们获取更多技术支持>>
参考文献
Heo SJ, Enriquez LD, Federman S, et al. Compact CRISPR genetic screens enabled by improved guide RNA library cloning. Genome Biol. 2024; 25(1): 25.
doi: 10.1186/s13059-023-03132-3.
 CRISPR基因编辑技术服务
CRISPR基因编辑技术服务
 红棉 · 基因敲除计划
红棉 · 基因敲除计划
































 联系电话
联系电话
 投诉电话
投诉电话
