IF=17.7|源井KO细胞为昆虫学研究注入新活力

IF=17.7|源井KO细胞为昆虫学研究注入新活力

研究背景
人们都说母亲是伟大的,她为了孩子的生长发育,呕心沥血。在自然界,绝大部分昆虫是没有抚育行为的。也就是说,当昆虫妈妈把自己的后代以产卵的形式留下之后,它们就静静地死去。那昆虫妈妈如何无微不至的照顾后代呢?有的昆虫是将自己的卵产在寄主植物上,卵一孵化就有食物。有的昆虫将卵产在土壤或隐蔽的环境中,以利于后代度过不良环境。
蝗虫作为群聚型昆虫,它们的母亲也为后代适应高种群密度做好充分准备。飞蝗受到高种群密度影响时,会改变后代卵的大小、数量、抗寒性、孵化特征等1-4,产生由种群密度诱导的跨代效应。其中,卵孵化同步性为群居型幼虫聚集、成虫交配及大规模迁飞奠定了基础。之前研究报道群居飞蝗卵巢中miR-276通过激活一个胚胎发育必需的辅转录因子Brahma (BRM)而诱导后代卵同步孵化的机制3。然而,高种群密度到生殖系统中miR-276的高表达的信号传递机制仍然未知,解析生殖系统小RNA响应外界环境信号机制将解决跨代效应研究中尚未回答的关键科学问题。
摘要
近日,中国科学院动物研究所康乐院士团队在Nature Communications上发表了题为“Parental experiences orchestrate locust egg hatching synchrony by regulating nuclear export of precursor miRNA”的研究论文。在这项工作中,以飞蝗具备的种群密度依赖的表型可塑性为模式,揭示了雌性飞蝗高种群密度经历通过生殖系统miRNA合成通路调控后代卵同步孵化的机制。其中,该研究采用了源井构建的敲除XPO5基因的293 T细胞以评估PTBP1是否能在独立于XPO5的情况下发挥作用。
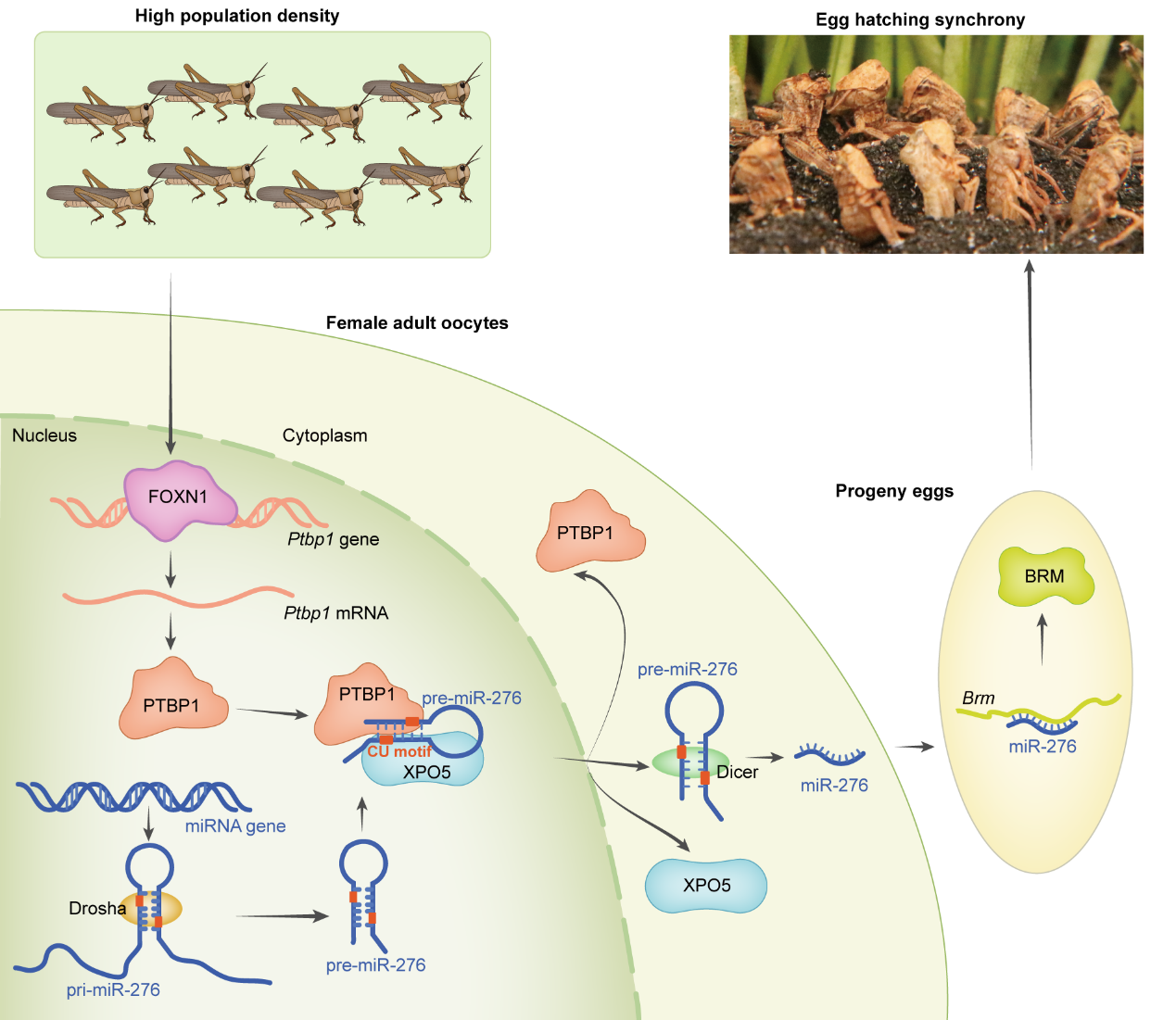
FOXN1-PTBP1–miR-276响应高种群密度调控后代卵同步孵化的作用模式
图文导读
首先,根据miRNA生物合成各个阶段,针对miR-276合成的中间产物设计特异性引物,并且在不含有本底miR-276的细胞系HEK 293T中证实飞蝗miR-276转录产物(pri-miR-276)以及前体(pre-miR-276)序列的正确性。通过比较群、散飞蝗卵母细胞里这些中间产物的表达量,发现群居飞蝗卵母细胞中miR-276高表达是由于pre-miR-276高效出核造成的(见图1)。
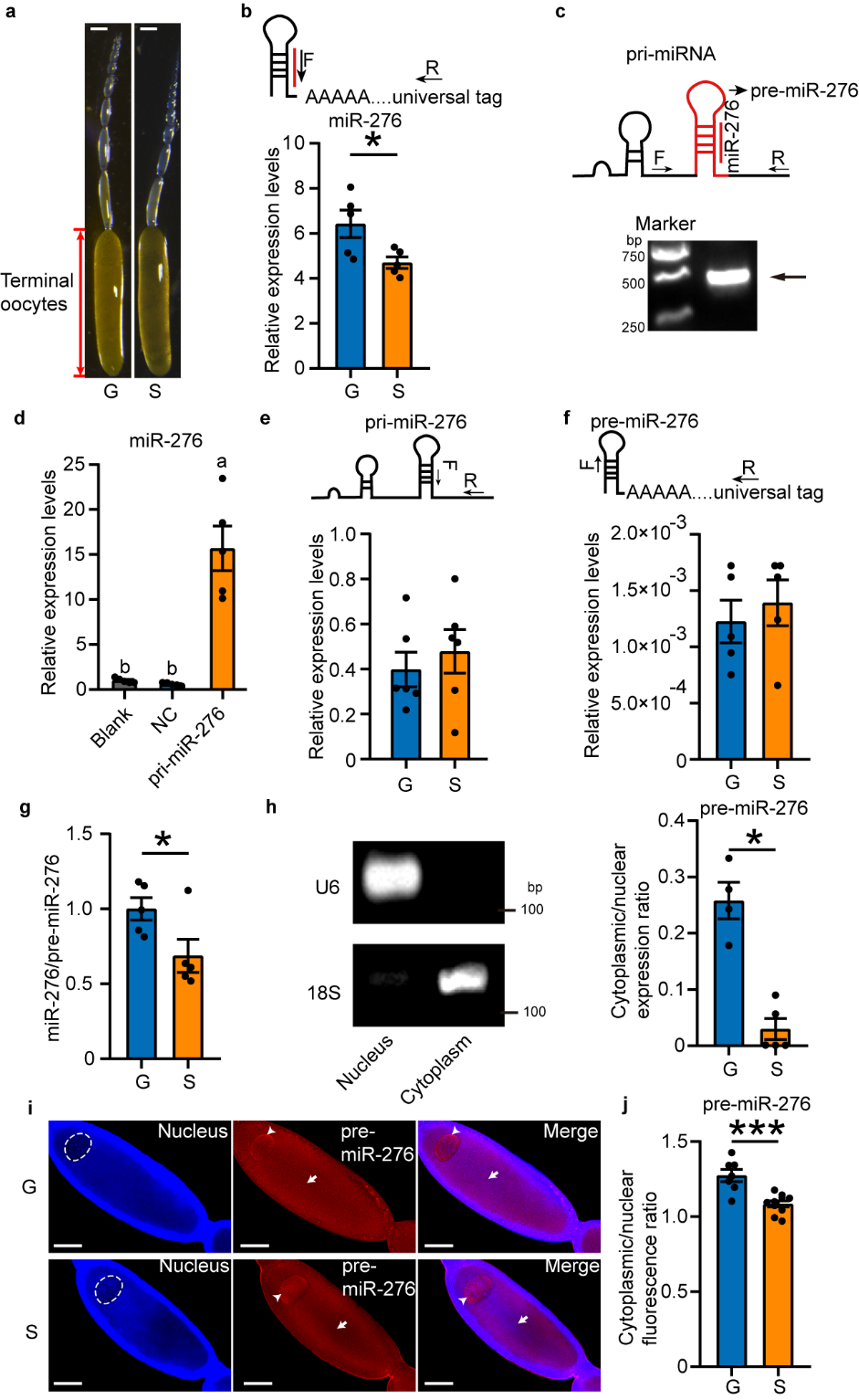
图1. 群居飞蝗末端卵母细胞中pre-miR-276出核效率高于散居飞蝗。
为进一步探究哪些蛋白因子参与pre-miR-276出核转运,研究人员通过设计pre-miR-276特异性探针,进行卵母细胞RNA pull-down实验拉取与pre-miR-276互作结合的蛋白,筛选出一些可能参与pre-miR-276出核转运的蛋白因子。并结合飞蝗体内干扰以及体外细胞过表达实验证实RNA结合蛋白-PTBP1促进pre-miR-276的出核转运(见图2)。
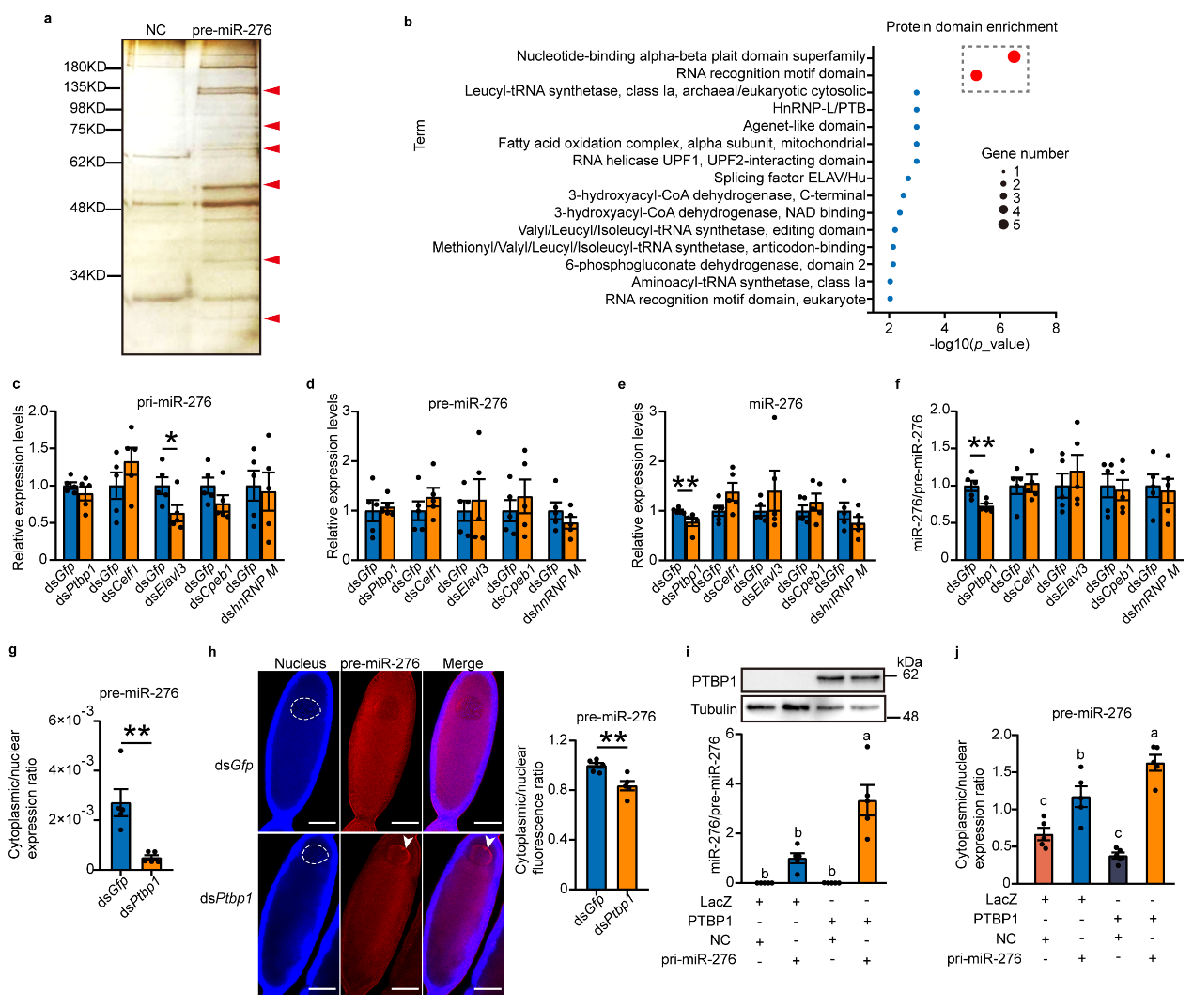
图2. PTBP1促进pre-miR-276出核转运。
由于PTBP1是一个RNA结合蛋白,通常位于信号通路下游,为研究PTBP1是如何响应外界高种群密度变化,该研究进一步寻找PTBP1上游响应种群密度信号的因子。通过定量发现,群散飞蝗Ptbp1在转录水平上响应种群密度变化。通过对Ptbp1的启动子扩增、验证并对Ptbp1的启动子进行预测筛选出5个可能的转录因子,然后通过体内干扰、EMSA实验、体外细胞启动子活性验证,证实FOXN1是Ptbp1的转录因子(见图3)。
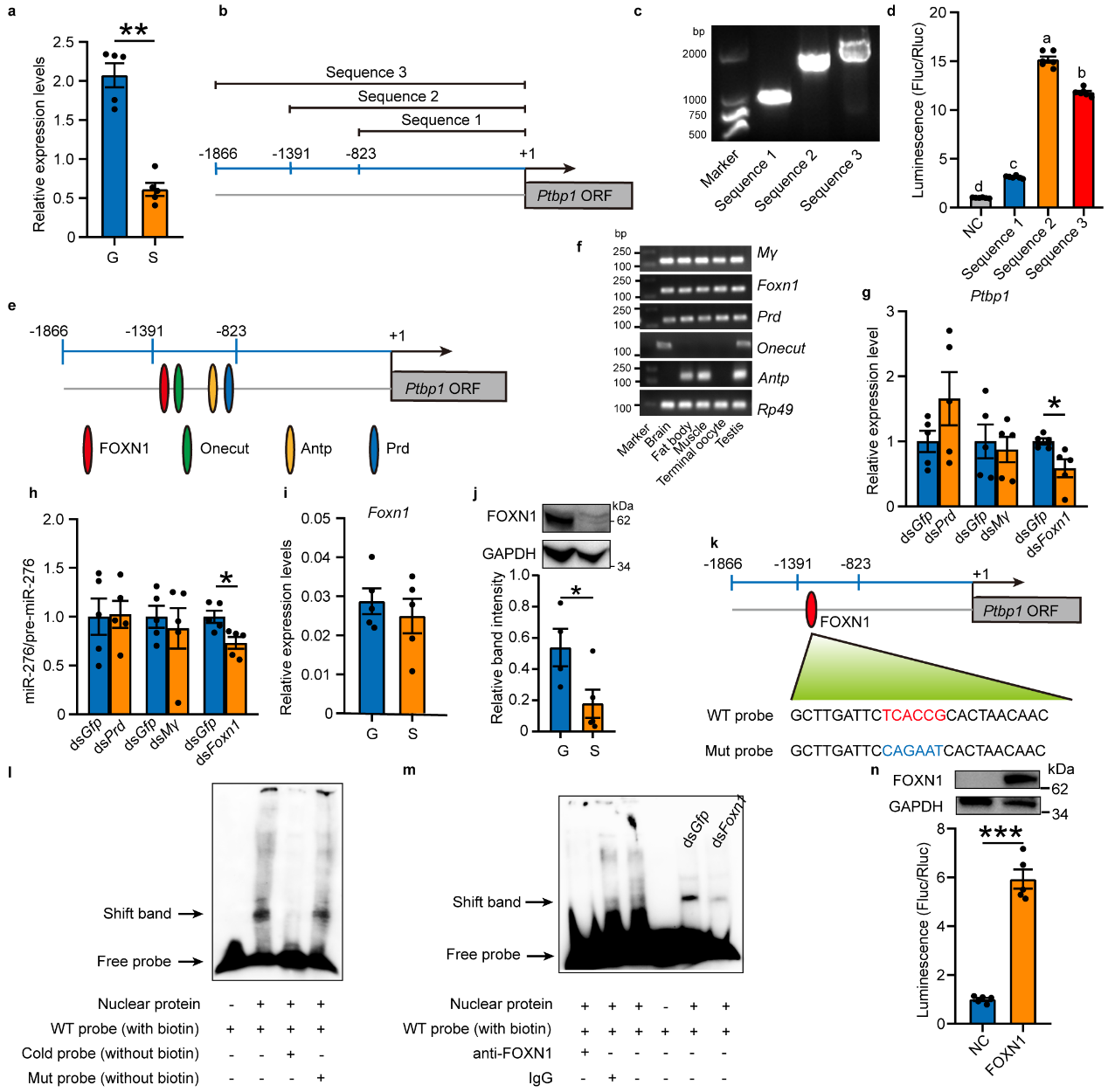
图3.转录因子FOXN1激活Ptbp1的转录。
为从机制上研究PTBP1是如何促进pre-miR-276的出核转运,该研究首先通过RNA-蛋白互作拉取实验证实PTBP1与pre-miR-276直接结合。飞蝗PTBP1是一个核质穿梭蛋白,PTBP1就可能是通过独立作用或者是与是pre-miRNA出核转运所必须的转运蛋白Exportin-5 (XPO5) 5互作来发挥作用。该研究采用了源井构建的敲除XPO5基因的293 T细胞以评估PTBP1是否能在独立于XPO5的情况下发挥作用。发现在缺失XPO5的情况下,PTBP1不能独立发挥功能。经过蛋白互作模型预测、体内外蛋白互作实验发现PTBP1能与XPO5互作。更为重要的是,体内干扰以及细胞过表达实验证明,PTBP1是通过与XPO5促进pre-miR-276出核转运(见图4)。
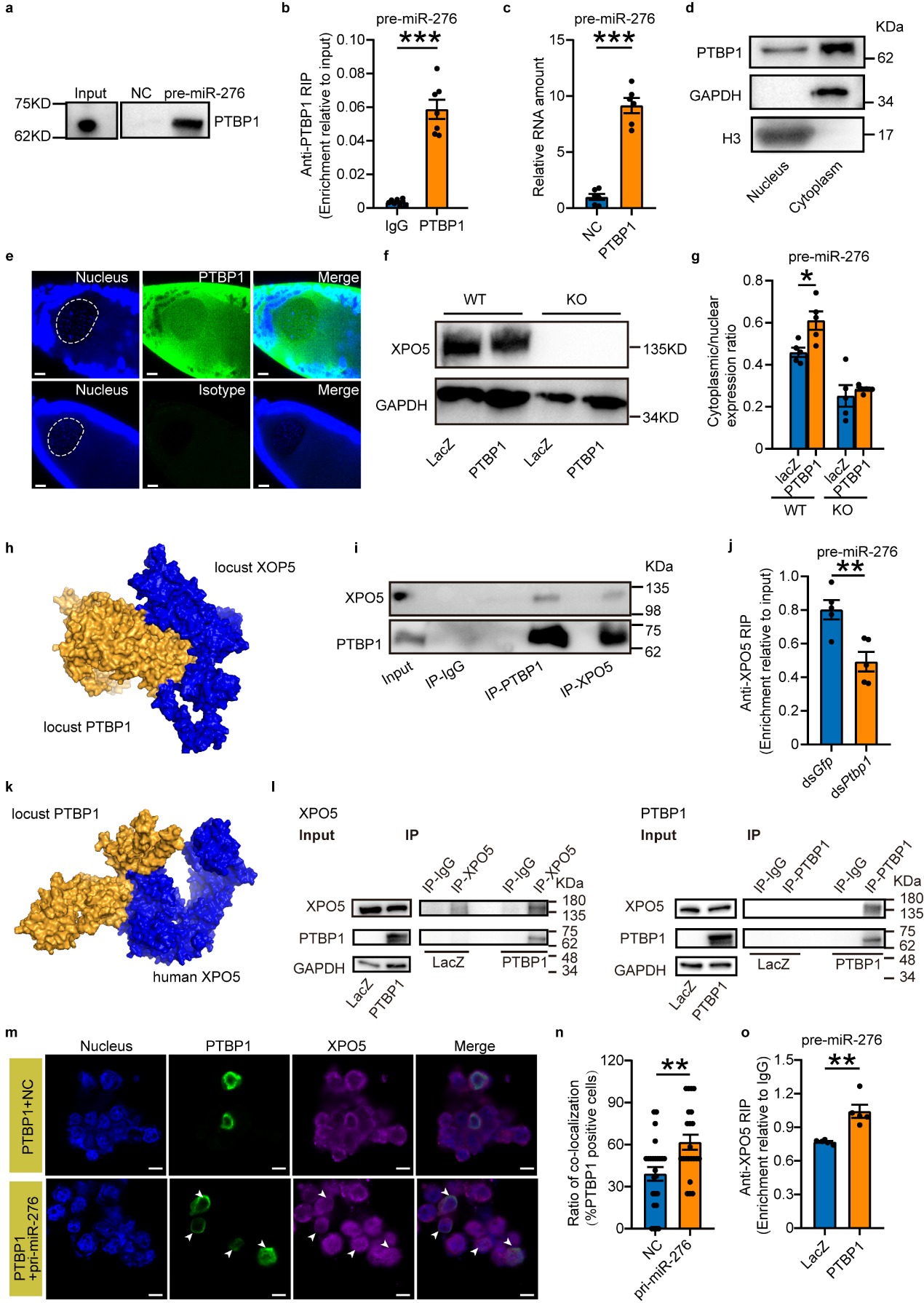
图4. PTBP1通过与XPO5互作促进pre-miR-276的出核转运。
PTBP1是如何识别pre-miR-276的呢?该研究首先猜测PTBP1与XPO5相同,即通过识别pre-miR-276普遍具有的二级茎环结构发挥功能。然而,当选取飞蝗卵巢中群居高表达的8个miRNA进行检测,发现干扰Ptbp1后发现不能抑制这些miRNA的前体(pre-miRNAs)的出核转运。说明PTBP1对miRNA前体出核作用并不是普遍的,即PTBP1不是通过识别pre-miRNA二级茎环结构发挥功能。由于PTBP1是多聚密度结合蛋白6,而pre-miR-276的序列在5p和3p链上各存在一个“CU motif”,正好与PTBP1结合的motif吻合。通过设计碱基突变实验发现,任意一个“CU motif”的突变后,PTBP1促进pre-miRNA出核功能不复存在,并且PTBP1促进pre-miRNA与XPO5结合的作用也消失,而非“CU motif”的突变并不影响PTBP1对pre-miRNA出核的促进作用。所以,这两个“CU motifs”对于PTBP1发挥作用缺一不可。所以,PTBP1是通过识别的“CU motifs”发挥促进pre-miR-276出核转运的功能(见图5)。
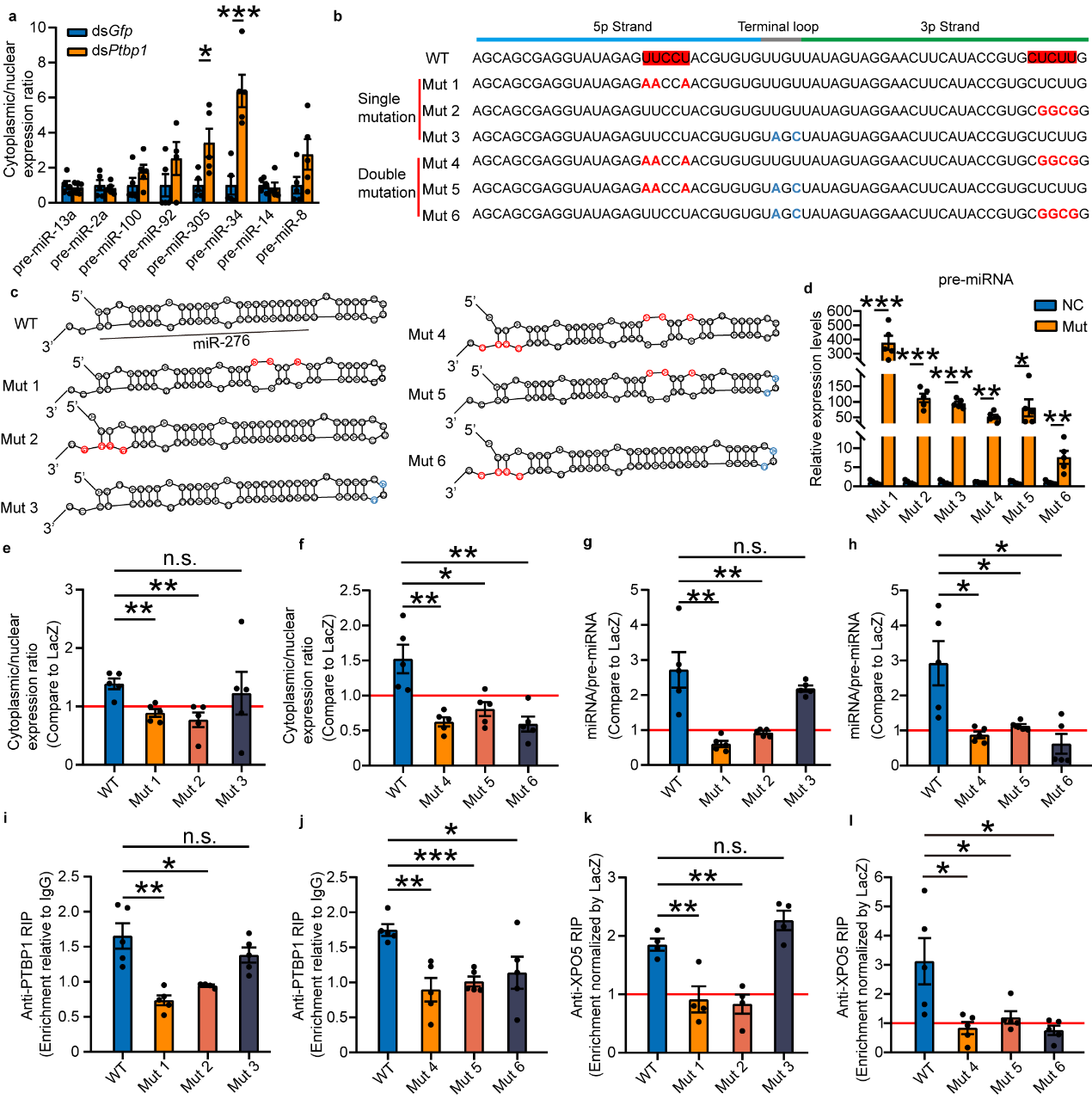
图5. PTBP1依赖pre-miR-276上的“CU motifs”促进pre-miR-276的出核转运。
之前研究发现,高种群密度经历的飞蝗通过激活miR-276高表达而促进飞蝗后代卵孵化同步性3。并且PTBP1、miR-276及靶标BRM在群居卵母细胞中高表达模式能够传递给后代卵。为进一步探究PTBP1是否也会通过影响miR-276的表达影响后代卵的孵化,通过雌虫体内干扰Ptbp1以及体内干扰Ptbp1后过表达miR-276进行表型回补,分析后代卵孵化时间发现雌虫生殖系统PTBP1能通过激活miR-276促进后代卵的同步孵化(见图6)。
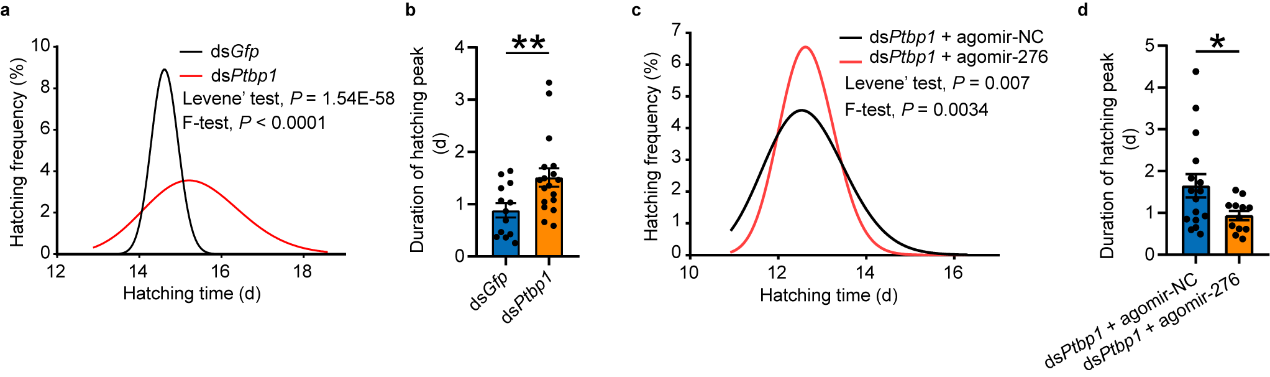
图6. PTBP1-miR-276促进飞蝗后代卵的同步孵化。
最后,该研究还探索了PTBP1介导的pre-miR-276出核转运机制的普遍性。因为miR-276是昆虫特有的miRNA,并且pre-miR-276尤其是“CU motifs”在昆虫中高度保守。该研究以果蝇为代表,验证PTBP1也能够促进pre-miR-276的出核转运,暗示了PTBP1发挥功能在昆虫中的保守性(见图7)。
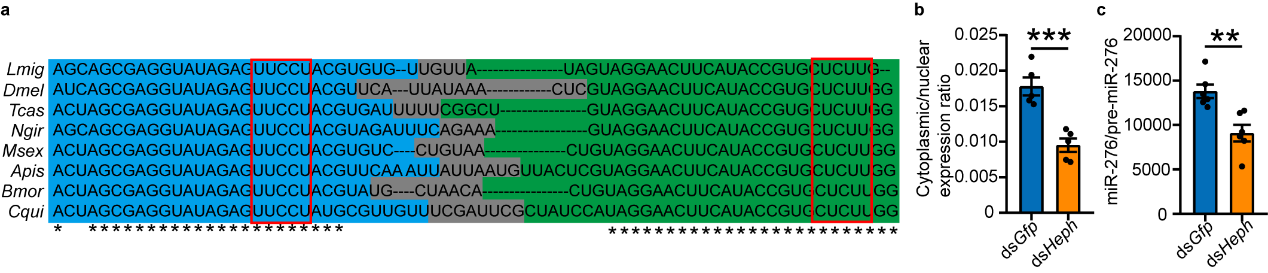
图7. 果蝇中PTBP1促进pre-miR-276的出核转运。
总结展望
该研究揭示了高种群密度经历下的飞蝗末端卵母细胞中转录因子FOXN1激活PTBP1,PTBP1通过识别pre-miR-276上的“标签”诱导PTBP1与之结合而招募更多的XPO5而促进pre-miR-276的高效出核导致miR-276高表达,miR-276-BRM诱导了后代卵同步孵化。并且PTBP1促进pre-miR-276出核的机制在昆虫中是保守的。这项研究将动物的生活经历与生物体内微观分子调控通路有机结合,提出了miRNA合成加工与如何响应环境的新思路和新见解,深化了对环境介导的跨代效应机制的理解。特别是提出飞蝗高种群密度经历调控后代卵同步孵化,为未来的幼虫聚集和成虫大规模迁飞奠定基础。所以,亲代根据自己的生活经历,预先安排后代的生活史对策,有利于后代适应母亲曾经生活过的环境。
参考文献:
1. Chen, Q. Q., He, J., Ma, C., Yu, D., Kang, L. Syntaxin 1A modulates the sexual maturity rate and progeny egg size related to phase changes in locusts. Insect Biochem. Mol. Biol. 56, 1-8 (2015).
2. Chen, B., et al. Paternal epigenetic effects of population density on locust phase-related characteristics associated with heat-shock protein expression. Mol. Ecol. 24, 851-862 (2015).
3. He, J., et al. MicroRNA-276 promotes egg-hatching synchrony by up-regulating brm in locusts. Proc. Natl Acad. Sci. USA 113, 584-589 (2016).
4. Wang, H. S., et al. Parental phase status affects the cold hardiness of progeny eggs in locusts. Funct. Ecol. 26, 379-389 (2012).
5. Lund, E., Güttinger, S., Calado, A., Dahlberg, J. E., Kutay, U. Nuclear export of microRNA precursors. Science 303, 95-98 (2004).
6. Han, A., et al. De novo prediction of
PTBP1 binding and splicing targets reveals unexpected features of its RNA recognition and function. PLoS
Comput. Biol. 10, e1003442 (2014).










