基因敲除HeLa细胞——医学突破的先锋

医学突破的先锋--CRISPR介导的HeLa细胞基因编辑

1952年,Henrietta Lack(HeLa)细胞成为第一个可以在实验室中无限传代的人类细胞系,而这些细胞被科学家们标记为“永生的”。研究人员最初是从侵袭性宫颈癌肿瘤中提取出的HeLa细胞。这些细胞在不断供应的营养物质下持续繁殖,并在不到24小时内生产出新一代的细胞。与许多肿瘤一样,Hela细胞具有错误组成的基因组,其中多个染色体有一个或多个拷贝:正常细胞有46条染色体,而Hela细胞包含76至80条染色体,其中一些染色体严重突变。同时,这些细胞还表达了过反应性端粒酶,即每次分裂后重建端粒,防止细胞衰老,以允许HeLa细胞无限传代。
HeLa细胞系为许多医学突破都做出了贡献,从研究太空零重力效应和脊髓灰质炎疫苗的开发,到白血病、艾滋病病毒和癌症的全球性研究。尽管现在有许多其他细胞系也在使用,但自HeLa细胞被分离以来,HeLa细胞在当今医学研究的大多数领域都取得了一定的进展,尤其是在癌症生物学,传染病,基础微生物学等领域都取得了一些重大进展。涉及HeLa细胞的研究已经在超过11000份科学刊物中描述过,其中包括三份获得过诺贝尔奖的:2008年发现人乳头瘤病毒(HPV)是宫颈癌的致病因子;2009年发现端粒酶可以保护染色体的端粒,从而防止染色体的降解,和2014年促进在细胞生长方面的实时观察。这个惊人的数字清晰地表明这些细胞在过去六十年中对研究的重要性。
HeLa细胞的多功能性和强大功能使其成为必不可少的实验室工具,继续为人类健康与疾病的基础提供新的线索。HeLa细胞是热门的细胞模型之一,也是生命科学家的工具,去研究疾病或治疗活性药物分子的作用机制,以及破译细胞信号的传导,如DNA损伤修复。研究人员选择HeLa细胞作为多学科研究的工具,因为它具有永生的细胞特征,可适用于多组学数据库(基因组学,蛋白质组学,和转录组学),并且易于利用这些数据库来设计和改进研究项目。最近的一项研究就是利用HeLa细胞研究SARS-CoV-2病毒在人体中的感染性。研究COVD-19的科学家们使用HeLa细胞以确定病毒用于进入人体细胞的受体。基于对HeLa细胞的研究,新型冠状病毒的研究取得了前所未有的进展。
应用范围:
HeLa细胞已经发展成为研究各种课题的有用模型。源自HPV转化的宫颈腺癌细胞的HeLa细胞已被用于各种研究,包括但不限于癌症的研究。作为一种肿瘤模型,它们已被广泛应用于研究中:
1. 肿瘤细胞的迁移和侵袭
2. 药物开发
3. 细胞死亡途径
CRISPR-U™ 基因编辑在HeLa细胞的应用
CRISPR-Cas系统已经成为了革命性的基因组编辑工具,为生命科学的发展和我们对生命的理解提供了巨大的动力。CRISPR系统用于精确的基因组编辑,可以通过用所需的供体序列取代靶基因序列来进行基因敲除或敲入基因组的操作。
HeLa细胞是一种侵袭性宫颈癌细胞系。在HeLa细胞系中进行基因编辑可以产生单个或多个基因敲除,正确的突变或插入报告基因转基因。这些经过基因组编辑的HeLa细胞系将提供给研究人员用于研究癌症,癌症治疗,细胞死亡,功能基因组学,信号通路,药物研发,药物反应和细胞治疗。
图:针对工程ESC和iPSC的CRISPR-U™定制流程
源井生物自主研发的CRISPR-U™可用于HeLa细胞系的基因操作。因此,利用CRISPR/Cas9系统可以在HeLa细胞中实现基因组编辑。源井生物可以定制真核HeLa细胞中的基因编辑,也可以在动物模型中进行各种基因改造。
源井生物自主研发的 CRISPR-U™ 技术优化了真核细胞和动物基因编辑载体和过程。效率和准确度比传统方法提高了10倍。马上联系我们,了解与您研究相关的服务吧!
基因敲除Hela的研究案例:
CRISPR/Cas9介导的TERT的紊乱可抑制肿瘤细胞的存活
端粒是真核细胞中维持染色体完整性的一种独特结构。哺乳动物的端粒长度主要受端粒酶的调控,而端粒酶是一种核糖核蛋白,由逆转录酶(TERT)和RNA亚基(TERC)组成。TERC在所有细胞中都有组成性表达,而TERT的表达则是在大多数成年体细胞中受到时间和空间上的调控。在体细胞中,TERT被灭活,因此端粒酶活性不会被检测到。而大多数肿瘤细胞通过激活TERT的机制来阻止进行性端粒侵蚀以实现增殖永生。因此,TERT失活被认为是一种很有前景的肿瘤治疗手段,研究人员利用CRISPR/Cas9介导的基因编辑系统来靶向癌细胞中的TERT基因。为此,他们选择了三种不同的癌细胞系:宫颈癌细胞系(HeLa)、胰腺癌细胞系(PANC1)和乳腺癌细胞系(SUM159),并设计了三个分别针对人类TERT基因外显子2(E2)、外显子4(E4)和外显子6(E6)的gRNAs(sg1、sg2和sg3)。研究人员进一步设计了另外两个靶向TERT外显子4(E4)侧翼内含子的gRNAs(sg4和sg5)来产生KO-TERT细胞。在单倍剂量不足的研究中,突变的TERT细胞端粒酶活性降低,端粒变短。与WTPE-Hela细胞相比,细胞增殖测量、细胞培养密度和更强的β-gal染色信号都显示出突变的TERT细胞产生了严重的细胞衰老。Annexin V和PI染色也提供了细胞凋亡的证据:突变的TERT细胞比WTPE-Hela细胞具有更高的凋亡率;在体外肿瘤细胞中缺乏TERT单链则导致细胞的生长迟缓和加速死亡。
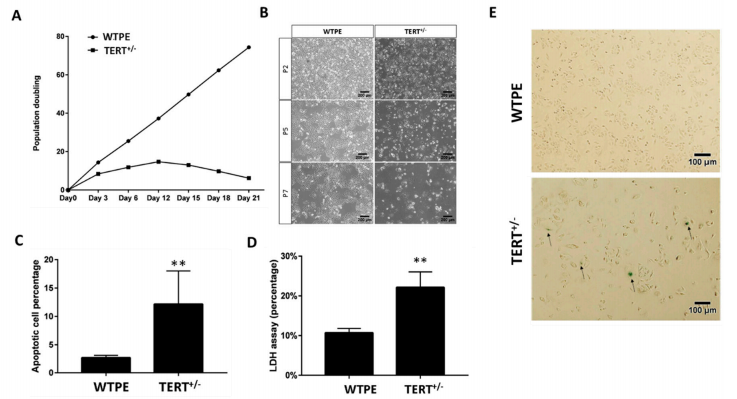
图1:Tert+/-癌细胞生长迟缓,细胞加速死亡。(A) WT和TERT+/-Hela细胞的群体倍增时间。(B) 第2代(P2)、P5和P7代WT和TERT+/-Hela细胞的光学显微镜图像。(C) WT和TERT+/-Hela细胞的β-gal染色。箭头指示严重衰老细胞的例子。(D) LDH法测定WT和TERT+/-Hela细胞的细胞死亡率。(E) 流式细胞术定量分析annexin-V和碘化丙啶(PI)染色对WT和TERT+/-Hela细胞中的细胞凋亡现象。**p<0.01。
研究人员进一步利用肿瘤异种移植的裸鼠模型研究了TERT单倍剂量不足对肿瘤细胞的存活率的影响,并在接种后6周检查异种移植物的大小。正如预期,WTPE-Hela细胞在动物体内生长成直径约2cm的肿瘤块,而TERT突变的Hela细胞在动物体内未能形成任何的肿瘤块,这一结果证明了Cas9介导的TERT单倍剂量不足可以有效地抑制肿瘤细胞的生长。
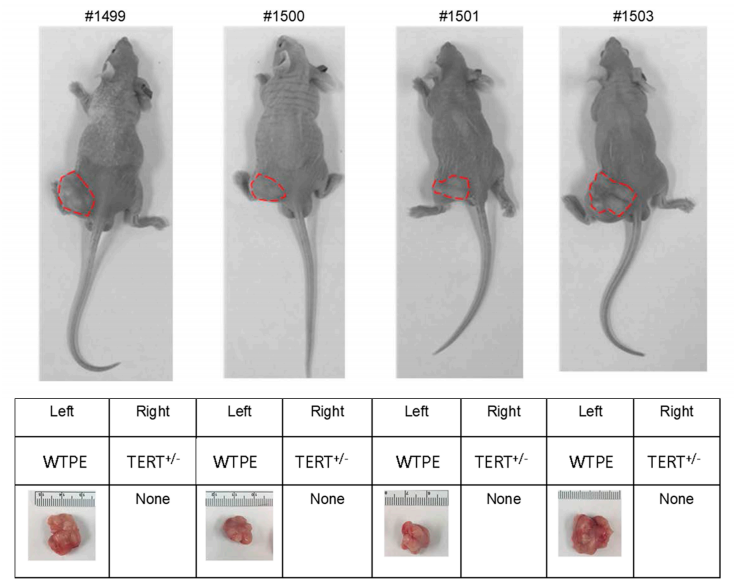
图2:WT和TERT+/-Hela细胞在裸鼠体内的异种移植
基因点突变Hela的研究案例:
利用CRISPR/Cas9引入致病性MSH2点突变导致人类细胞差异性基因组的失稳
重复去稳定化与人类疾病有多种关联,特别是在肿瘤性疾病中。微卫星不稳定性(MSI)被认为是简单反映了DNA错配修复(MMR)缺陷。而MSI+表型在各种人类肿瘤中并不相同。根据微卫星变化的频率,MSI分为MSI-H(高)和MSI-L(低)。此外,MSI还对其不同的定性模式进行了分类,如A型和B型。早前的一项研究表明,MMR基因敲除小鼠中的肿瘤并未表现出MSI-H肿瘤中典型的剧烈微卫星变化(即B型),而是小而细微的改变(即A型)。
林奇综合征(LS)被称为遗传性非息肉性结直肠癌(HNPCC),是遗传性结直肠癌(结肠癌)的最常见原因。MLHL,MSH2,MSH6,PMS2和EPCAM这些基因的突变是造成林奇综合征的因素。在这项研究中,利用CRISPR/Cas9系统将林奇综合征(LS)中的MSH2突变引入到HeLa细胞中。研究人员使用基因组编辑载体pSpCas9(BB)-2A-Puro(pX459),gRNA(由CRISPR-direct和CRISPR设计工具设计)并使用BbsI位点亚克隆到pX459中。使用Lipofectamine 2000将编码gRNA的pX459质粒和三种不同的单链ssODN共同转染到细胞中。产生的突变克隆清楚地表现出具有烷化剂耐受性和突变频率升高的MMR缺陷表型。然而,微卫星并没有像MSI-H肿瘤在林奇综合征患者中表现得那么显著地不稳定,且所有观察到的变化均为A型,而这恰恰证实了小鼠的结果。这些发现表明人类基因组中重复不稳定的分子机制是更加复杂的。
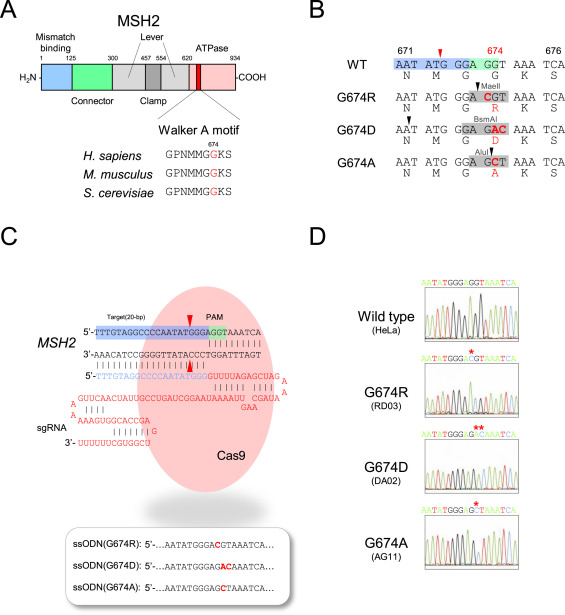
图:CRISPR/Cas9系统将MSH2 G674突变引入HeLa细胞。(A) 人MSH2蛋白中的功能区域,包括Walker A基序,(B)每个G674突变体版本中Walker A基序中的局部DNA和氨基酸序列,(C)用于实验的向导RNA和ssODN的结构。(D) Sanger方法确认每个已建立的HeLa克隆。
这项研究已成功获取了MSH2突变的HeLa克隆,并且这些已建立的克隆明显表现出具有烷化剂耐受性和升高的突变频率的MMR缺陷表型。尽管在这些克隆中,微卫星并未像林奇综合征患者中的肿瘤那样表现出明显不稳定。目前的发现表明,除了MMR缺陷外,以前无法识别出的分子机制可能是人类细胞重复失稳的根本。
基因敲入Hela的研究案例:
sgRNA的可逆遮盖/揭开策略(Cloaking/Uncloaking strategy)可控制CRISPR–Cas9在体外和活细胞中的基因编辑效率
RNA“遮盖”(RNA Cloaking)是控制RNA杂交,折叠和酶促相互作用的温和且可逆的一种化学方法。在这项研究中,研究人员使用叠氮化物取代的酰基咪唑试剂(NAI-N3)在合成后酰化RNA的20-OH基团,导致RNA折叠,杂交和功能的阻断。使用水溶性磷化氢处理后,此类聚酰化(“遮盖”)RNA的体外活性可被有效修复,这种水溶性磷化氢引起叠氮化物的施陶丁格还原反应,然后自发地失去酰基(“揭开”)。研究人员用sgRNAs研究这些可逆的遮盖/揭开现象,来调控CRISPR-Cas9系统在体外和活细胞中的基因编辑效率。
对于体外研究,研究人员设计了一种体外基因编辑模型平台,该平台具有103个核苷酸长(nt)的合成sgRNA,是以Cy5标记的双链DNA(dsDNA)为靶点并编码一部分的绿色荧光蛋白(GFP)。他们发现,当被“遮盖”的sgRNA与三羟丙基膦(THPP)或二苯基膦苯-3-磺酸盐(TPPMS)在浓度为1-5 mM的磷酸盐缓冲盐水(PBS)缓冲液中于37℃培养1小时时,得到了最高程度的Cas9介导的DNA切割——表现为在定量层面上活性被完全恢复到未处理sgRNA的水平。这种惊人的体外表现表明了该方法在CRISPR-Cas9诊断应用中的控制时机和启动方面是有潜在用途的。
对于体内研究,研究人员使用了GFP阳性的HeLa细胞,而这些细胞最初是通过核转染或商用阳离子脂质转染GFP靶向sgRNA和Cas9蛋白。他们进行了一项实验,基于与体外实验相同的步骤,用来测试“遮盖”由CART递送的GFP靶向sgRNA是否能够阻断在GFP阳性HeLa细胞中CRISPR–Cas9的基因组编辑。他们观察到用“遮盖”的sgRNA/Cas9 mRNA转染的GFP阳性细胞与未处理的细胞有着相同的GFP荧光表达水平。这证实了sgRNA的酰化基本上阻断了活细胞中的所有sgRNA的活性。因此,该研究强调了可逆RNA酰化可作为一种新的方法来控制基因组编辑的功能。
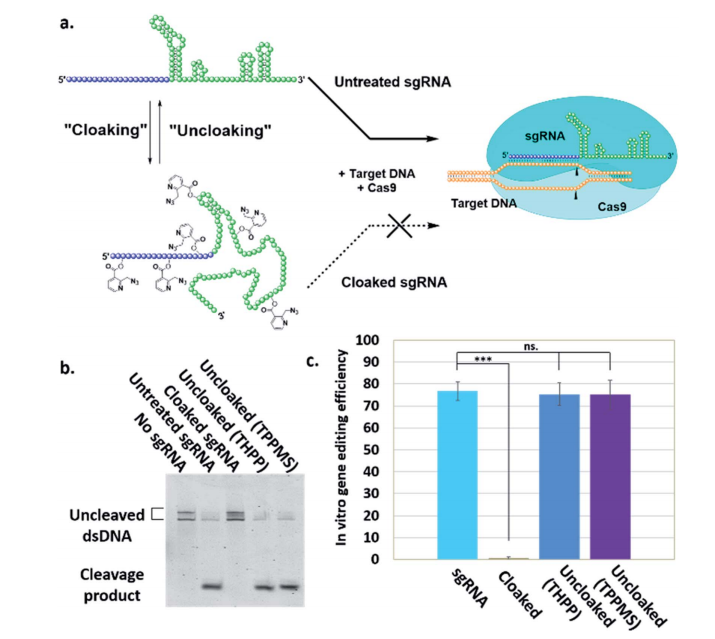
图1:(a)NAI-N3抑制CRISPR-Cas9基因编辑的机制。sgRNA的“遮盖”是通过Cas9核酸酶抑制RNA引导的DNA双链切割(b)使用Cy5标记的dsDNA和未处理的,“遮盖”的和“揭开、未遮盖”的(磷化氢处理的)sgRNA在体外的Cas9核酸酶测定的PAGE分析。(c) 条形图表示gRNA与Cas9s培养后的DNA裂解分数。
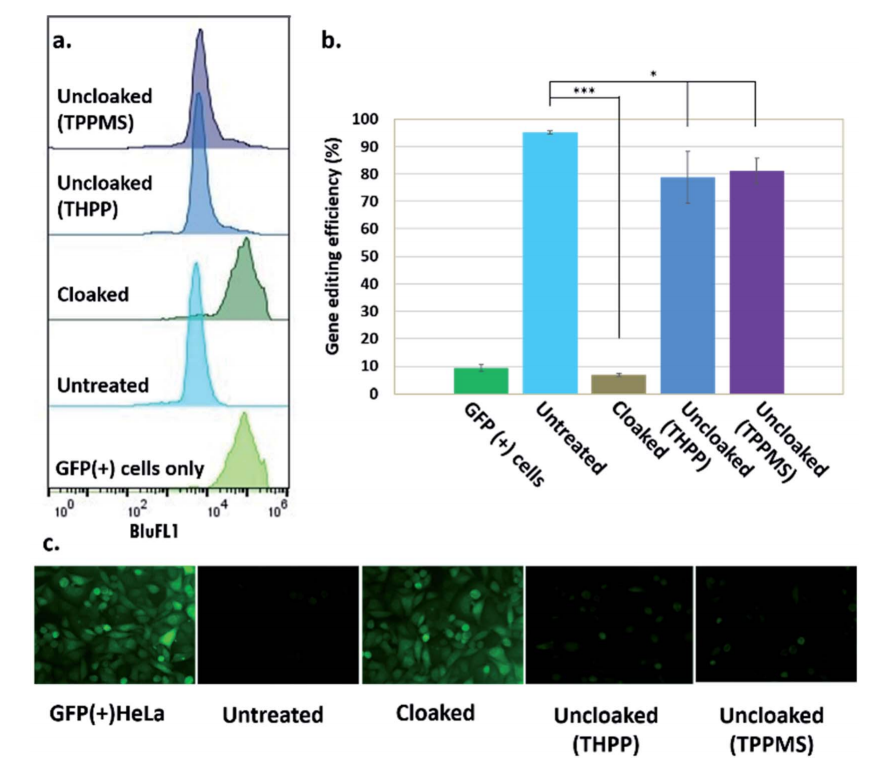
图2:磷化氢控制人类细胞中被“遮盖”的CRISPR–Cas9活性。(a) GFP阳性HeLa细胞的流式细胞仪分析显示,被“遮盖”的sgRNA活性丧失,并通过磷化氢处理修复编辑;(b)图表(数据来自流式细胞仪)显示未处理的,“遮盖”的和磷化氢处理的sgRNA的基因编辑效率(c)荧光显微镜图像显示用未处理的sgRNA和Cas9 mRNA转染后的GFP敲除,被“遮盖”的sgRNA失去编辑的作用,使GFP荧光不受影响,以及用磷化氢处理后细胞的编辑作用恢复。
参考文献:
CRISPR/Cas9-Mediated TERT Disruption in Cancer Cells. Int. J. Mol. Sci. 2020, 21, 653.
Differential genomic de-stabilization in human cells with pathogenic MSH2 mutations introduced by genome editing. Experimental Cell Research, 2019 (377) 24–35.
Reversible RNA acylation for control of CRISPR-Cas9 gene editing. Chem. Sci.,










