一文带你了解肺癌研究常用细胞模型!

一文带你了解肺癌研究常用细胞模型!
肺癌是全球发病率和死亡率最高的癌症之一,根据其组织病理学特点主要分为非小细胞癌(包括腺癌和鳞癌,占所有肺癌84%)为了深入理解肺癌的发展过程并探索有效的治疗手段,研究人员通常会利用肺癌细胞模型在体外模拟肺癌的生物学特性,为肺癌的诊断和治疗提供重要的科学依据。今天,小源给大家介绍一下肺癌研究中常用的细胞模型及其应用。
人非小细胞肺癌细胞(A549)
货号:YC-C016
来源:人肺腺癌细胞系,取自58岁男性的肺腺癌组织。
基因突变:KRAS突变(G12S)。
特性:该细胞系能分泌肺表面活性物质,具有Ⅱ型肺泡上皮细胞功能,是肺腺癌和Ⅱ型肺泡上皮细胞的经典模型[1]。广泛应用于抗癌药物筛选与评估、肺癌肿瘤、基因编辑等研究。
应用实例:
UXS1基因在细胞代谢等过程中可能有着重要作用。Doshi等人[2]使用UXS1-iKO(诱导型)A549细胞和UXS1-KOA549细胞,发现UXS1基因缺失会扰乱糖核苷酸的清除。与正常A549细胞相比,UXS1基因敲除的细胞在特定代谢条件下,增殖能力明显下降,显示出糖核苷酸代谢在维持癌细胞增殖优势中的关键作用。进一步研究发现糖核苷酸代谢的改变还影响了癌细胞的信号通路。一些与细胞存活和耐药相关的信号受到干扰,意味着通过调控糖核苷酸代谢,有可能克服癌细胞的耐药性,这为开发新型抗癌药物和联合治疗方案提供了新的思路。
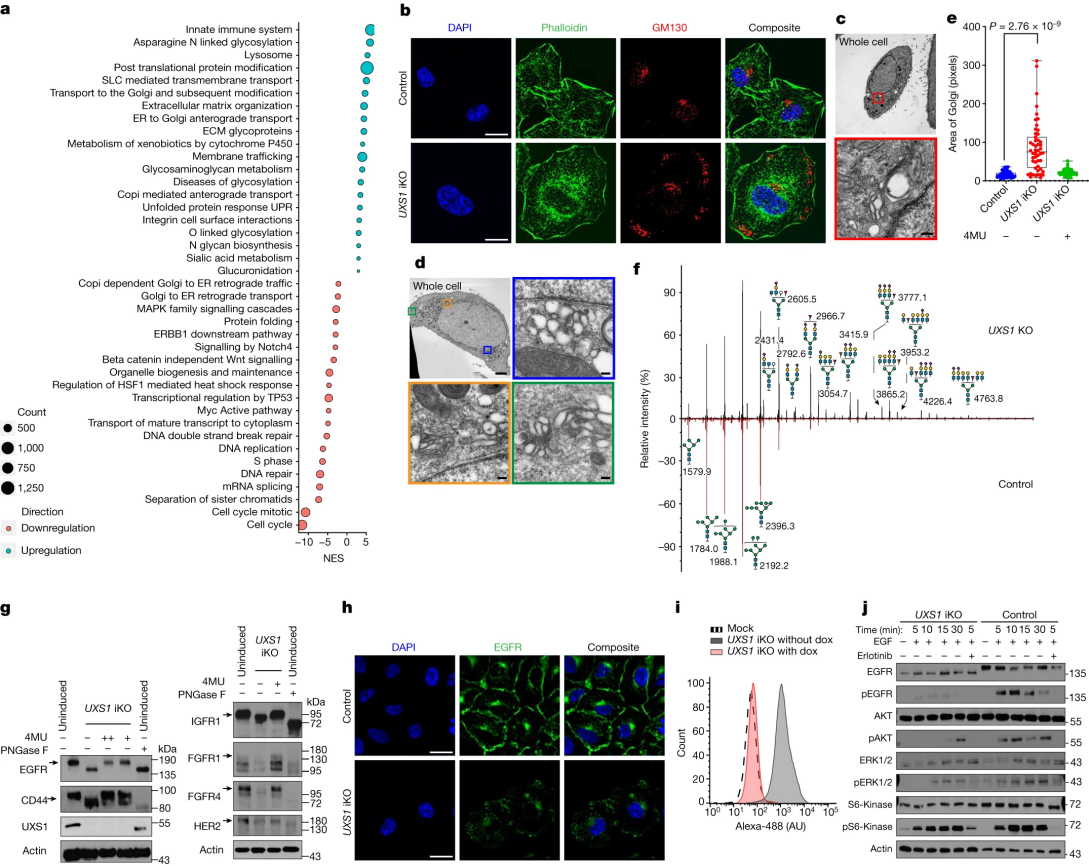
UDPGA 积累导致高尔基体功能障碍,从而导致 EGFR 失活。
人非小细胞肺癌细胞(NCI-H1299)
货号:YC-D010
来源:该细胞系是从接受过放射治疗的患者的肺淋巴结转移中建立的,属于非小细胞癌的大细胞癌亚型。
基因突变:TP53功能缺失[3]。
特性:TP53缺失导致对DNA损伤反应和修复能力异常。增殖能力、迁移和侵袭能力较强。常用于转移机制与肿瘤侵袭研究、TP53相关信号通路、药物耐药和靶向治疗研究等。
应用实例:
Yan等人[4]使用LAPTM4B基因敲除NCI-H1299细胞模型来研究LAPTM4B在非小细胞肺癌中对铁死亡的影响。研究发现,敲除LAPTM4B后,NCI-H1299细胞中与铁死亡相关的代谢物显著改变,特别是与谷胱甘肽代谢、半胱氨酸和蛋氨酸代谢相关。LAPTM4B的缺失导致细胞内脂质过氧化和丙二醛(MDA)水平增加,同时谷胱甘肽(GSH)水平下降,这些变化是铁死亡的典型特征。此外,LAPTM4B的缺失还导致线粒体结构异常,表现为线粒体体积缩小和变形。这些结果表明LAPTM4B通过抑制SLC7A11蛋白的泛素-蛋白酶体降解来抑制铁死亡,进而促进NSCLC的进展。
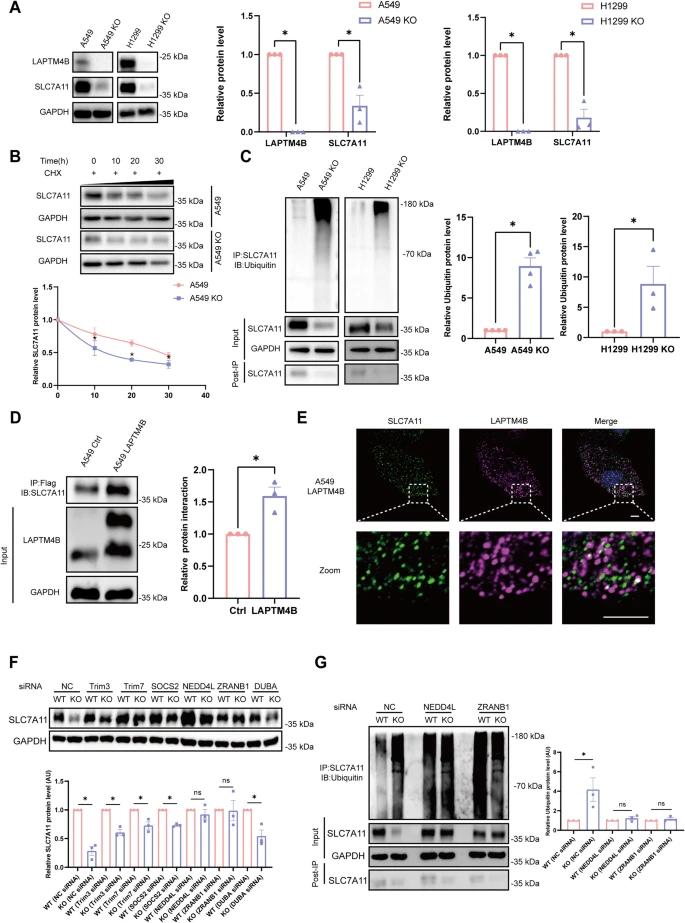
LAPTM4B抑制SLC7A11的泛素化-蛋白酶体降解。
人肺腺癌细胞(PC9)
货号:YC-C114
来源:肺腺癌(分化型)组织。
基因突变:EGFR突变(19号外显子缺失,del E746_A750)。
特性:EGFR突变使其对EGFR抑制剂高度敏感,是肺腺癌中经典的靶向治疗研究模型,尤其是EGFR-TKI(如厄洛替尼、奥希替尼)耐药性研究[5]。此外,还常应用于肿瘤增殖、侵袭机制等研究。
应用实例:
Pfeifer等人[6]使用了PC9细胞进行了全基因组CRISPR敲除(CRISPRn)和激活(CRISPRa)筛选,以识别EGFR突变肺癌对EGFR抑制剂耐药性的驱动因素。为了验证这些耐药基因,作者构建基因敲除或过表达细胞模型。其中,NF2基因的敲除导致PC9细胞对osimertinib产生耐药性,并激活了Hippo信号通路,特别是YAP1/WWTR1/TEAD轴。这一激活伴随着YAP1和WWTR1核定位的增加以及TEAD报告基因活性的增强。此外,抑制YAP1/WWTR1/TEAD轴的转录活性能够重新敏感化耐药细胞对osimertinib的反应,并预防耐药性持久细胞的形成。这些结果表明,Hippo信号通路在EGFR突变肺癌的耐药性中起着关键作用,为克服耐药性提供了新的治疗靶点。
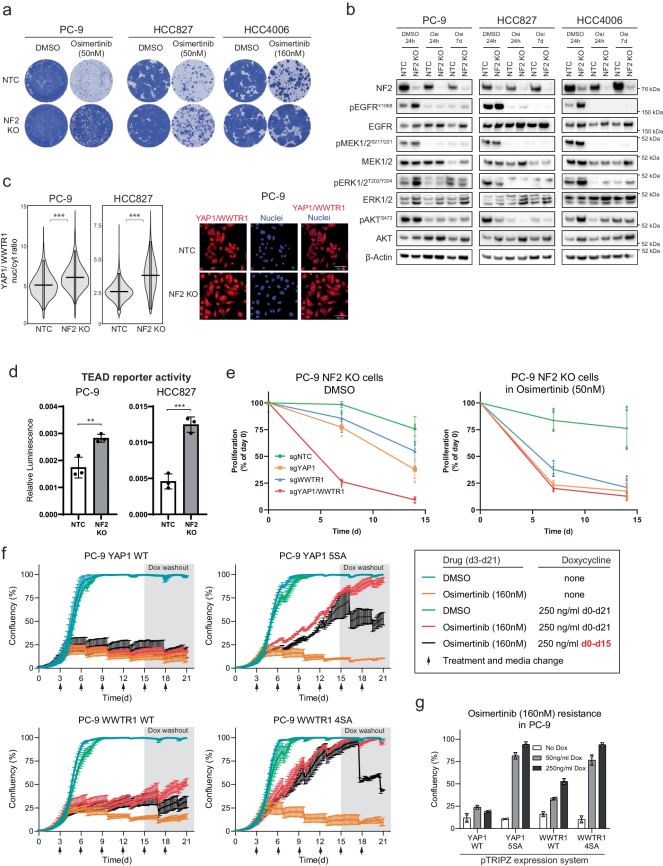
Hippo 通路调节 EGFR 突变肺癌中的奥希替尼反应
小结
除了上述常见的肺癌细胞,还有NCI-H520(人肺鳞癌细胞)、NCI-H1688(人小细胞肺癌细胞)、LLC(小鼠肺癌细胞)等不同类型的肺癌细胞,它们独特的起源、基因突变特征、耐药性、转移能力等,使其成为肺癌研究中的重要工具。大家可以根据不同的研究目标,选择合适的细胞系有助于更有效地推动肺癌的基础和临床研究。
参考文献:
[1]Foster, Kimberly A., et al. "Characterization of the A549 cell line as a type II pulmonary epithelial cell model for drug metabolism." Experimental cell research 243.2 (1998): 359-366.
[2]Doshi, M.B., Lee, N., Tseyang, T. et al. Disruption of sugar nucleotide clearance is a therapeutic vulnerability of cancer cells. Nature 623, 625–632 (2023). https://doi.org/10.1038/s41586-023-06676-3
[3]Maj, Ewa, et al. "Differential response of lung cancer cell lines to vitamin D derivatives depending on EGFR, KRAS, p53 mutation status and VDR polymorphism." The Journal of steroid biochemistry and molecular biology 193 (2019): 105431.
[4]Yan, R., Liu, D., Guo, H. et al. LAPTM4B counteracts ferroptosis via suppressing the ubiquitin-proteasome degradation of SLC7A11 in non-small cell lung cancer. Cell Death Dis 15, 436 (2024). https://doi.org/10.1038/s41419-024-06836-x
[5]Ohara, Shuta, Kenichi Suda, and Tetsuya Mitsudomi. "Cell Line Models for Acquired Resistance to First-Line Osimertinib in Lung Cancers—Applications and Limitations." Cells 10.2 (2021): 354.
[6]Pfeifer, M., Brammeld, J.S., Price, S. et al. Genome-wide CRISPR screens identify the YAP/TEAD axis as a driver of persister cells in EGFR mutant lung cancer. Commun Biol 7, 497 (2024). https://doi.org/10.1038/s42003-024-06190-w










