文献解读:如何利用激活型筛选平台发现耐药突变

耐药性的出现与发展在癌症的靶向治疗过程中是几乎不可避免的。基因突变是导致耐药的常见原因之一,在经药物治疗的患者群体中识别耐药突变位点往往需要耗费大量时间与金钱成本。近期发表在《Cell Reports》上的一篇名为《Accelerated drug-resistant variant discovery with an enhanced, scalable mutagenic base editor platform》的文章开发了新的药筛平台,该平台基于对胞嘧啶碱基编辑器BE4的修饰改造,从而得到了条件性激活诱导胞苷脱氨酶(activation-induced cytidine deaminase , AID)-dCas9(AID-dCas9)系统。AID-dCas9系统不但展现出极高的编辑效率,而且有更广的编辑范围与更丰富的编辑异质性,更是在识别、分类和功能注释耐药突变等方面展现出极大的应用潜力,该系统的问世为发现顺式与反式突变、分析蛋白功能关系提供了简单且具扩展性的研究手段。
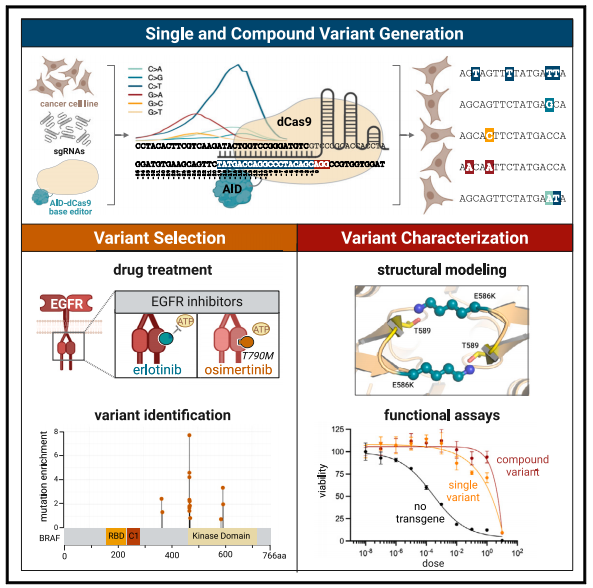
图一 全文摘要图
AID-dCas9系统的高效多样化编辑效果
该系统基于常用碱基编辑器BE4的原有结构进行了以下改造:首先,将rAPOBEC1替换为直接融合的超活性AID(AID*delta)胞苷脱氨酶;与APOBEC1相比,AID具有更宽的编辑窗口,但仍保持高编辑效率和以C>T编辑为主的特异性。其次,通过移除与BE4串联的尿嘧啶DNA糖基化酶抑制剂(uracil DNA glycosylase inhibitor,UGI)结构域阻止尿嘧啶切除,并促进易错DNA修复通路与突变多样性;在缺乏UGI结构域时,AID-Cas9n的编辑特异性不再局限于C>T,还包括C>A和C>G编辑。最后,使用dCas9而非Cas9n以避免切口酶活性干扰。与Cas9n相比,使用dCas9在有效避免DNA断裂、减少Indels的同时,使编辑类型更为丰富;虽然整体编辑效率较AID-Cas9n降低,但在sgRNA靶序列5'端附近(-28至-19 bp PAM位点)观察到G>A、G>C和G>T编辑显著增加。此外,除导致单碱基突变之外,AID-dCas9系统可通过单次编辑产生复合变异,并且能结合相应测序手段识别到这些不同类型的突变,兼顾了cis/trans效应。
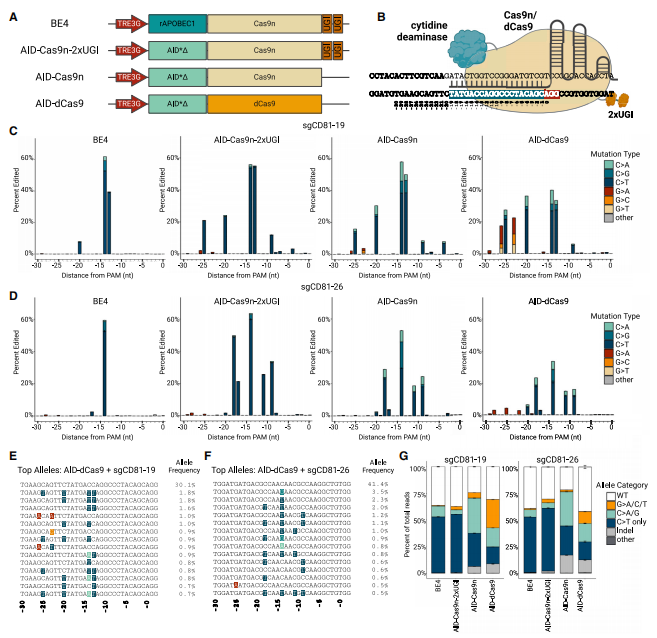
图二 高诱变碱基编辑器AID-dCas9的优化与表征
AID-dCas9系统的靶向编辑
为测试AID-dCas9系统的靶向编辑能力,针对CD81五号外显子的140 bp区域(CD81ex5)设计21个sgRNA,使它们密集覆盖整个靶区域;单个sgRNA以慢病毒为载体感染PC-9细胞,多西环素(DOX)处理6天后再通过测序分析编辑效果。此外,为控制每个碱基编辑器在基因组中的暴露时间和持续时间,我们将单个碱基编辑器构建在DOX诱导型启动子下游的piggyBac转座子中。结果显示,虽然大部分C>N编辑集中在PAM位点上游-21至-12 bp处,但G>N编辑则富集在距离PAM位点上游-28至-19 bp处,编辑窗口比BE4大大加宽;CD81扩增子所有核苷酸中的约45%和约80%的GC核苷酸可被AID-dCas9编辑,显示其编辑范围之广。
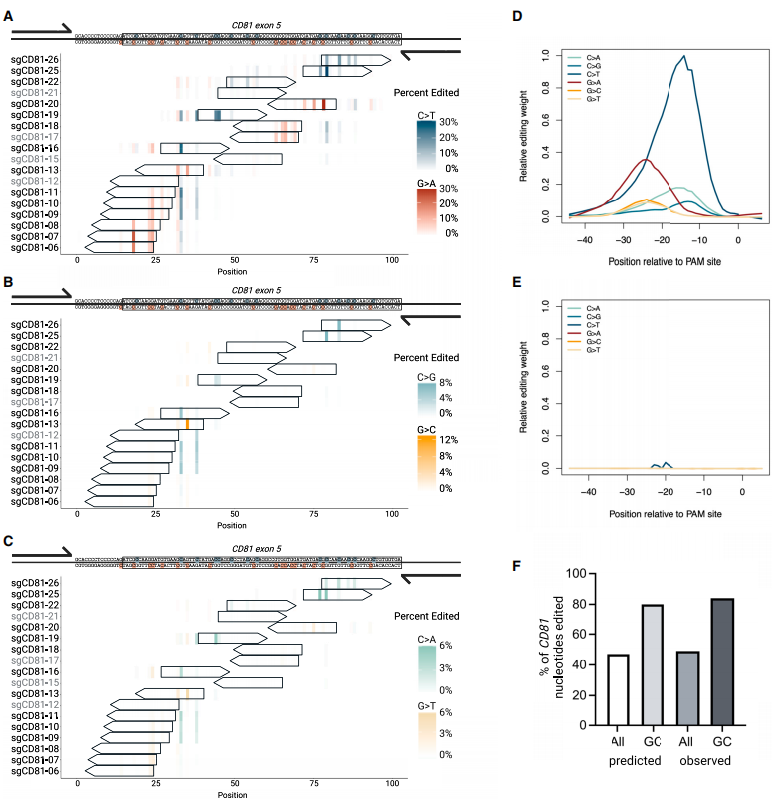
图三 靶向AID-dCas9与平铺覆盖式sgRNAs联合实现广泛突变
应用AID-dCas9系统进行激活型耐药突变筛选
在证明AID-dCas9系统能够在给定区间诱发高密度和多样化突变后,研究人员进一步评估了该系统在实际药物筛选背景下生成耐药突变的能力。为此,他们基于非小细胞肺癌(non-small cell lung cancer,NSCLC)中EGFR抑制剂耐药机制建立了一系列筛选和后续验证:用上述平铺式覆盖思路设计了一个靶向EGFR和BRAF整个编码区所有潜在NGG PAM位点的sgRNA文库,并用以同种方式设计非MAPK通路基因ESR1上的sgRNA文库和50个非靶向sgRNA作为阴性对照。将对应的sgRNA文库导入PC9细胞后,通过DOX诱导耐药突变产生;经不同剂量厄洛替尼/奥希替尼或DMSO处理后收集存活细胞并进行测序,分析sgRNA的富集情况。
对厄洛替尼与奥希替尼两种药物均以高低两个药物剂量分别进行筛选,通过低剂量富集更大范围的耐药突变,以防漏筛;通过高剂量富集更强效、更具穿透性的耐药变异。此外,研究人员还测试了DOX的作用天数对sgRNA富集是否存在明显差异。结果显示,与耐药相关的sgRNA出现明显富集:EGFR四个区域和BRAF两个区域的sgRNA富集具有高度显著性,与EGFR区域2重叠的显著富集sgRNA(对应T790突变位点)在所有高剂量奥希替尼组以外的3组中都普遍存在,而BRAF区域1(对应G479突变位点)中的sgRNA在所有高剂量厄洛替尼组以外的3组中均富集。对厄洛替尼和低剂量奥希替尼组收集的细胞进行靶向基因组测序,发现存在T790M位点突变,并且在该位点相对于其他等位基因占比较高;在厄洛替尼和奥希替尼的低剂量组中,T790M与L792F突变顺式共存。同时,对BRAF区域1的测序揭示了BRAF G469A和T470I两个位点不但产生单点变异,而且产生二者间顺式共存的突变。因此,应用AID-dCas9系统可成功生成已知临床耐药突变,以上结果验证了该平台的可靠性。
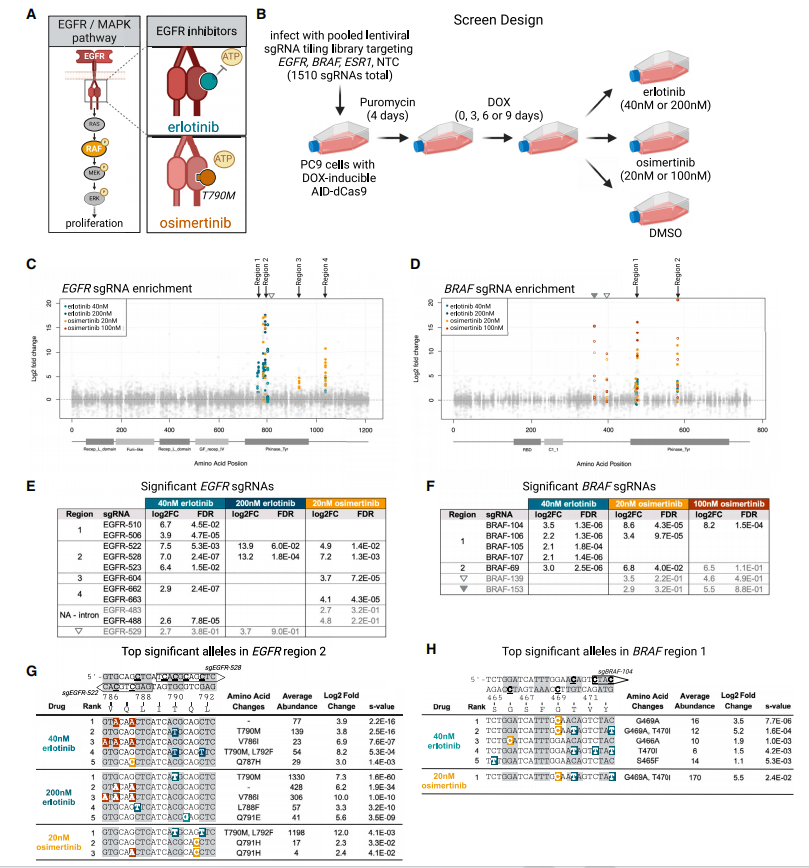
图四 混合池式碱基编辑筛选EGFR抑制剂耐药性
为将AID-dCas9系统编辑后的厄洛替尼/奥希替尼耐药变异体进行颗粒度统计,研究人员结合单细胞测序,对上一阶段筛选出的sgRNAs进行了二次筛选,并使用能够评估基因组编辑结果的Tapestri平台进行基因分型。首先针对上述6个强突变区(EGFR1-4区与BRAF1-2区)、4个弱突变区以及1个靶向内含子连接区(证明通过编码区突变导致耐药性出现)与1个阴性对照区设计了1个sgRNA混合文库,并在将其构建至载体上后导入AID-dCase-PC-9细胞。表达碱基编辑器的细胞经亚文库转染后,依次接受嘌呤霉素处理、DOX诱导,再按照厄洛替尼或奥希替尼的IC95剂量给药,最后通过单细胞测序检测每个sgRNA靶向的位点。每组测序样本约4,000个细胞,每个扩增子约测得20,000-200,000个reads。对于EGFR中显著富集的变异,氨基酸突变主要发生在厄洛替尼组别并聚集在EGFR激酶结构域;其中T790M和邻近的Q791E突变较为显著,L792F和其他T790近端突变也有较高富集。对单细胞数据进行汇总与伪批量分析,以确定这些突变的等位基因:在厄洛替尼组中,按s值排名的EGFR中前三个显著富集的等位基因均包含T790M突变(T790M、T790M/Q791E和T790M/L792F),这说明该突变是耐药的主要驱动因素;在奥希替尼组中只筛选到T790M/L792F一组显著等位基因,这与前人的研究结果“L792F等额外突变与T790M顺式存在才能赋予对该分子耐药性”是一致的。
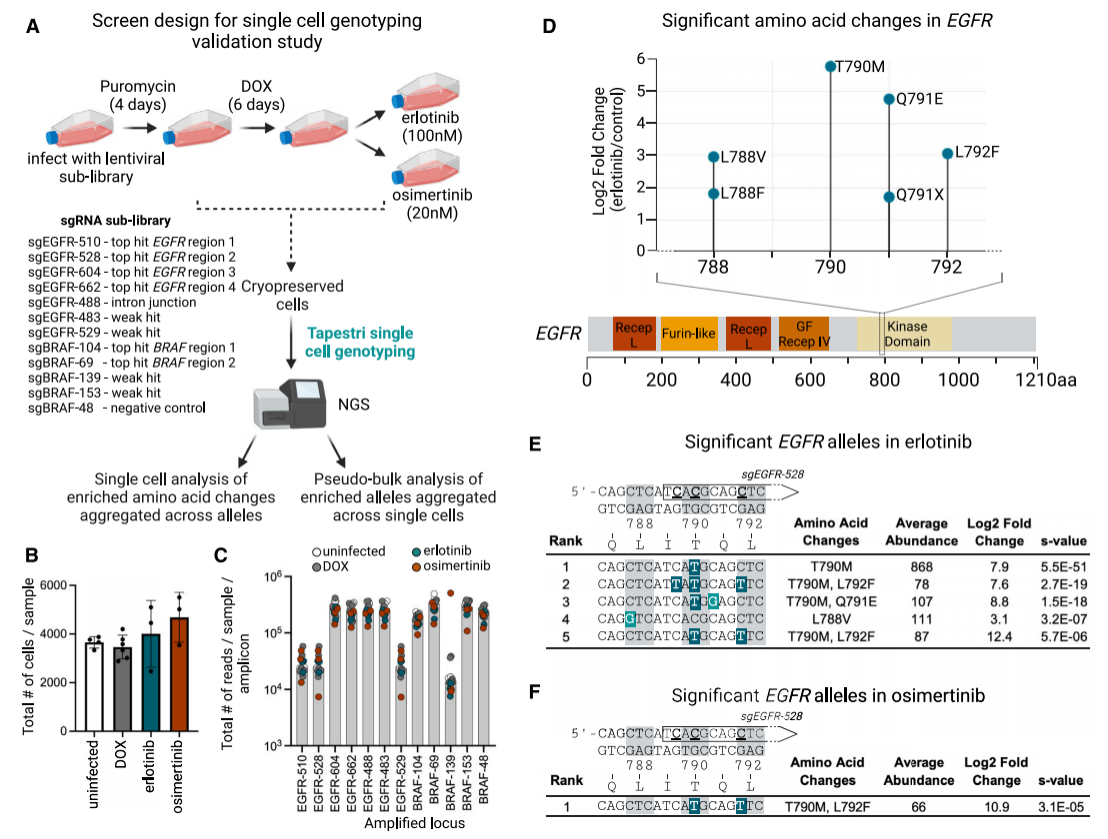
图五 对EGFR突变进行单细胞基因分型以确定其对EGFR抑制剂的耐药性
对BRAF中单个氨基酸突变进行分析,结果显示奥希替尼组中三个具有富集变异的区域,其中包括最为重要且已被广泛研究的G469位点,它的单点变异与复合变异均被显著富集;经奥希替尼和厄洛替尼处理后,G469A等位基因与T470I或T470K顺式共存,且G469/T470的复合变异是本研究通过AID-dCas9系统筛选出的全新变异类型。
在BRAF激酶结构域中观察到跨越E586-D594序列的另一组显著富集突变,该区域包含了II类BRAF突变体E586K和III类突变体D594N;同样地,E586K/D594N单点或复合变异在EGFR小分子抑制剂背景下的耐药作用此前尚未被揭示。此外,两个编码A366P变异的等位基因在奥希替尼组中显著富集。鉴于以上位点的耐药突变均未被广泛研究,作者将蛋白质结构进行模拟以对上述突变建模,用表型与靶点结构相关联的方式解释耐药机制。
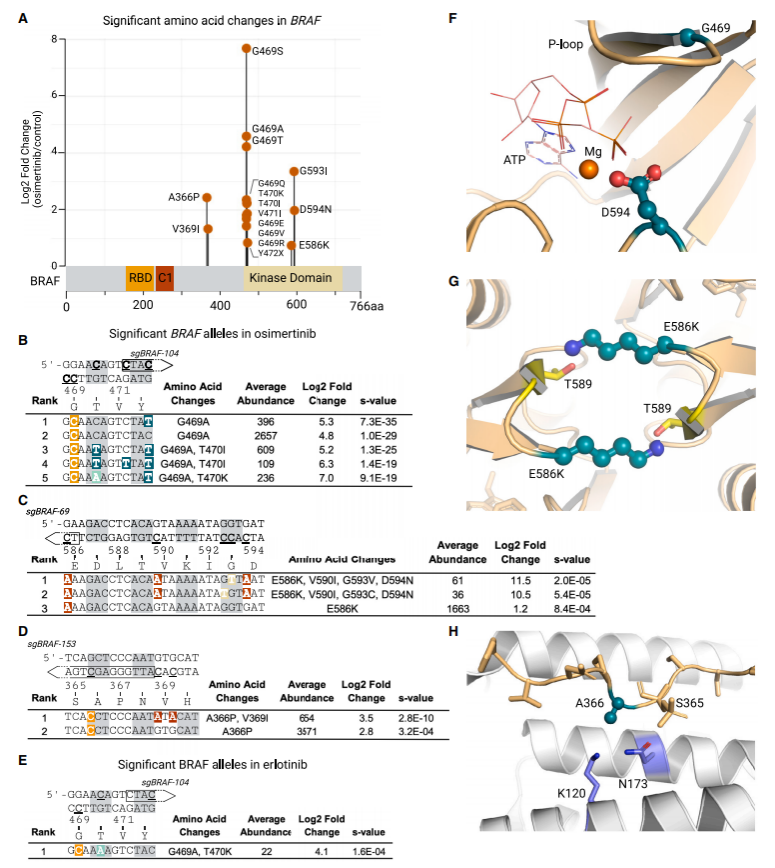
图六 对EGFR抑制剂处理后富集的BRAF变异进行单细胞基因分型的数据
在找出潜在耐药突变位点后,研究人员通过互补性过表达实验对这些突变(尤其是BRAF相关突变)进行了验证:将一系列突变在DOX诱导型PC-9细胞模型中过表达,并使用含有激活外显子19缺失(del746-750)的EGFR突变作为阳性对照。结果显示,T790M单点突变(厄洛替尼组别)和T790M/L792F复合突变(厄洛替尼与奥希替尼组别)都能够协同外显子19缺失突变增强耐药性。在BRAF过表达后也能够观察到不同种类或剂量药物作用下的耐药性显著改善现象:虽然BRAF G469A单点突变能够导致细胞对厄洛替尼和奥希替尼的耐药性,但它的两种复合突变并未增强这种效应;相比之下,E586K/D594N/G593V/V590I复合突变的存在显著提高了厄洛替尼与奥希替尼跨剂量反应的耐药性,也高于E586K单点突变导致的耐药性。同样地,BRAF A366P单点突变也能在一定程度上增强细胞对厄洛替尼与奥希替尼的耐药性。对携带DOX诱导型突变EGFR或BRAF转基因的细胞系进行MAPK通路激活标志物的蛋白质印迹分析,包括磷酸化EGFR、磷酸化ERK和磷酸化MEK,检测条件涵盖存在或不存在DOX、厄洛替尼和奥希替尼的情况。此外,western blot结果显示,在EGFR抑制剂(厄洛替尼/奥希替尼)处理下,对照组中的pMEK/pERK(EGFR通路中的下游调控因子)表达被显著抑制,意味着通路关闭;在BRAF突变组中pMEK/pERK维持高表达,说明通路被持续激活。由此得出,BRAF突变体能不经过EGFR维持EGFR/MAPK的下游信号通路,从而增强耐药性,使细胞存活。
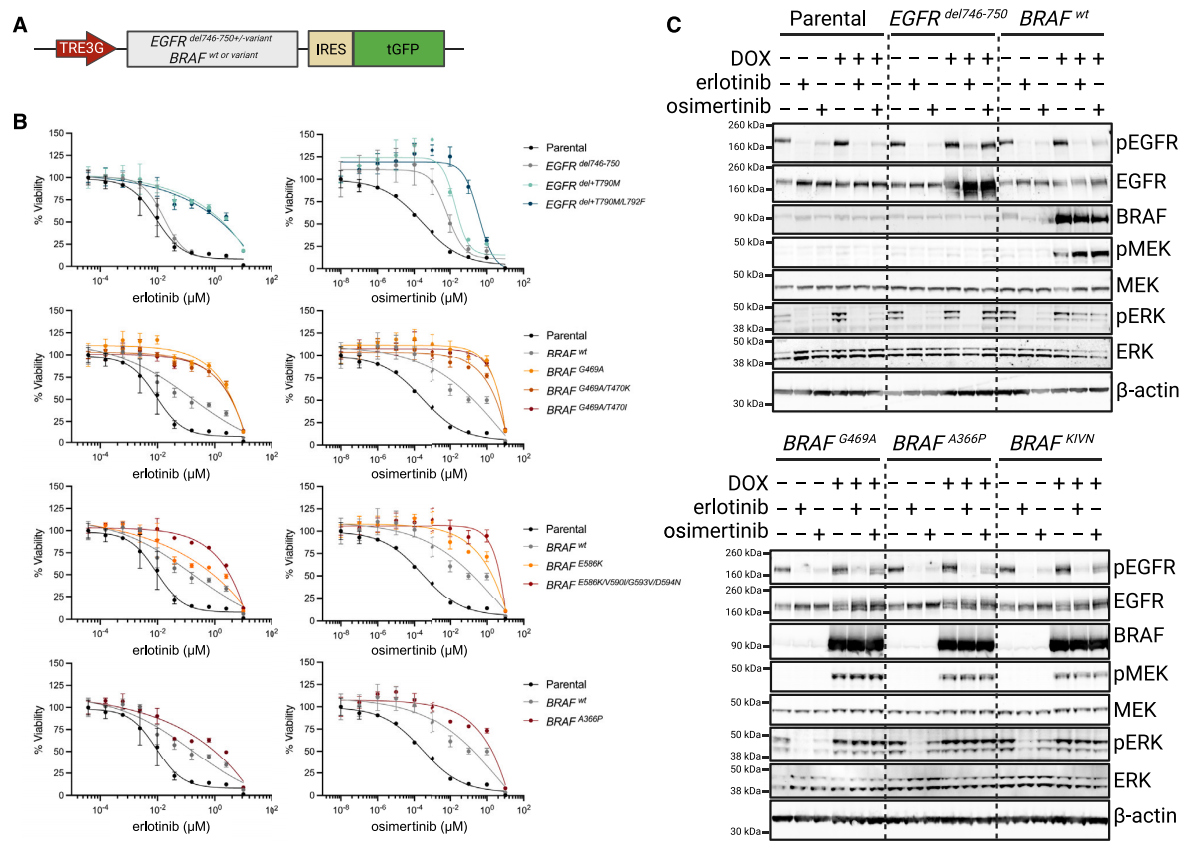
图七 DOX诱导型EGFR和BRAF突变基因的过表达重现了EGFR抑制剂耐药表型
总结
这篇文章的核心在于开发了一种高效诱变的碱基编辑器,本文中开发的AID-dCas9系统通过以下三个关键创新实现了多样化突变:
-
1) 用超活性AID替代APOBEC1以拓宽编辑窗口;
-
2) 去除UGI结构域,从而允许错误修复,增加突变类型;
-
3) 使用dCas9避免切口酶活性干扰。经此改造后的碱基编辑平台能在靶位点产生C>N和G>N多种替换,编辑效率相较改造前显著增加。
本文的整体实验设计是完善且循序渐进的:先在模型基因CD81上成功验证编辑多样性,再将其应用到在实际药物筛选背景下,以评估AID-dCas9系统生成耐药突变的能力。在筛选过程中采用分级策略,先用平铺式设计思路针对相关信号通路设计sgRNA文库进行初筛并抓取到sgRNA富集区域,以增强效率和缩小范围,再用着重覆盖上述突变富集区并结合单细胞基因分型进行高精度筛选,最终找到潜在耐药突变位点。此外,本文在sgRNA文库设计与对照选择、药物剂量分层筛选、编辑-筛选时间窗口优化等方面的设计十分巧妙,将其与高覆盖度测序相结合,能够有效避免弱突变的漏筛。这套思路不但可以应用于肿瘤耐药相关的研究,而且在探寻与疾病表型相关的基因或通路、发现增强细胞治疗或免疫治疗相关靶点、识别促进细胞命运转变的相关因子、搜索增强病毒感染性或宿主抵抗病毒的防御因子等研究领域中也同样可以借鉴。
无论您是探索未知机制、筛选药物靶点、破解耐药难题,还是优化免疫治疗策略,源井生物始终是您值得信赖的科研伙伴。我们提供全方位的CRISPR功能筛选解决方案,涵盖个性化方案设计(结合科研问题定制筛选策略)、高效稳定的实验执行(依托成熟平台确保数据质量)、全流程无缝衔接(覆盖CRISPR文库筛选各环节)以及专业的数据解读支持(助您从海量数据中提炼生物学洞见)。如果您正在寻找一站式、高质量的功能筛选服务,欢迎联系我们,开启精准科研的新篇章!
参考文献
Dorighi KM, Zhu A, Fortin JP, Hung-Hao Lo J, Sudhamsu J, Wendorff TJ, Durinck S, Callow M, Foster SA, Haley B. Accelerated drug-resistant variant discovery with an enhanced, scalable mutagenic base editor platform. Cell Rep. 2024 Jun 25;43(6): 114313. doi: 10.1016/j.celrep.2024.114313.
 CRISPR基因编辑技术服务
CRISPR基因编辑技术服务
 红棉 · 基因敲除计划
红棉 · 基因敲除计划






























 联系电话
联系电话
 投诉电话
投诉电话
