非增殖差异的表型,能用CRISPR文库筛选靶点吗?

非增殖差异的表型,能用CRISPR文库筛选靶点吗?

作为功能基因组靶点筛选神器,CRISPR文库的风头可谓一时无两。最近,小源收到很多朋友对文库筛选靶点遇到的同一困惑:是不是只有跟细胞存活、增殖凋亡有关的表型才能用文库来筛靶点?否则目的细胞如何被富集呢?
当然,能出现细胞增殖凋亡差异的表型确实更容易做CRISPR文库筛选,但是非增殖差异的表型也是有解决办法滴。通过流式细胞分选,可以将存在抗体结合差异(膜表面蛋白)或荧光表达差异(报告细胞系)的细胞进行分选富集,再利用NGS分析目标靶点。
今天小源给大家分享一篇Ryan J. Park等人在Nature Genetics(IF=30.8)上发表的文章:A genome-wide CRISPR screen identifies restricted a set of HIV host dependency factors ,该文章就是借助报告细胞和流式分选,用CRISPR文库筛选到HIV病毒感染相关的宿主因子[1]。
HIV病毒是一种攻击人体免疫系统细胞的慢病毒,属于逆转录病毒,其主要攻击目标是人体免疫系统中最重要的CD4+ T淋巴细胞,HIV病毒通过大量破坏CD4+ T细胞使人体丧失免疫功能,从而引发艾滋病(AIDS),严重威胁人类的生命健康。
图1 HIV病毒概述图(来源于百度词条)
目前,HIV治疗药物主要是直接靶向病毒蛋白,这类药物在临床使用中易产生获得性耐药突变。与直接靶向病毒蛋白药物相比,针对HIV感染所需的宿主蛋白的药物在抗HIV治疗中能提高耐药屏障,是一种更为理想的HIV治疗方法,对于阐明HIV突破宿主细胞防御系统的分子机制、开发HIV治疗新靶点,具有非常重大的理论意义和实际应用价值。
CCR5(C-C趋化因子受体5)是R5噬性HIV-1(CCR5-tropic HIV-1)感染宿主的辅助受体,与CD4协同作用,通过结合HIV病毒糖蛋白gp120,促使HIV病毒进入宿主细胞。
在早期的利用RNAi筛选HIV治疗靶点的研究中,得到了近千个候选宿主因子,然而结果存在重叠性低、基因沉默不完全、脱靶效率高等缺点。在这篇文章中,作者利用全基因组CRISPR敲除文库筛选的方法,验证了原代CD4+ T细胞中噬CCR5 HIV毒株的5个相关宿主因子,分别为HIV感染所必需的共受体CD4和CCR5;在细胞外酪氨酸残基上硫酸化CCR5的共同途径中起作用,促进CCR5被HIV包膜识别的TPST2和SLC35B2;以及介导细胞间HIV传播的ALCAM基因,以上基因被敲除后均不影响细胞的存活,但能促使细胞抵抗HIV感染,这表明HIV依赖于有限数量的非必需宿主蛋白进行复制。同时,该结果也为HIV治疗提供了潜在靶点,为预防和治疗艾滋病提供了新的理念和途径。
由此可见,文库筛选的方法相比于RNAi具有更高的灵敏度和特异性,其中TPST2、SLC35B2和ALCAM这三个基因在早期的RNAi的研究中并未被筛选出来。
以下是该文章采用CRISPR文库进行筛选的流程介绍。
1、构建生理相关的CD4+ T细胞系模型
作者选择CD4+ T细胞白血病系CCRF-CEM细胞,在CCR5-hygro和HIV-1 LTR-GFP稳定表达的CCRF- CEM细胞上感染Cas9病毒,构建筛选HIV感染靶点的工具细胞株(CXRCas9细胞)(图2)。该细胞在HIV-1感染前高表达CCR5、低表达GFP,但是在HIV-1感染后,病毒产物的表达增加了HIV-1 LTR启动子的转录,导致GFP的高水平表达,从而允许在单细胞水平上通过流式细胞荧光分选技术检测细胞感染状态。
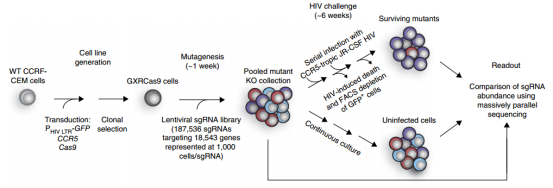
图2 HIV宿主依赖因子的全基因组CRISPR筛选
2、sgRNA文库制备与病毒包装
用文库包装成慢病毒之后感染CXRCas9细胞进行全基因组筛选,文库包含187536条sgRNA,靶向18543个蛋白编码基因(平均每个基因有10条sgRNA)和1504条非靶向对照sgRNA,感染24小时后用嘌呤霉素筛选出转染成功的细胞。
文库病毒感染1周后,用HIV病毒株JR-CSF以infect MOI为0.025去感染这些缺失不同受体的T细胞,感染1周后即可检测到GFP阳性细胞(~10-20%)。再过2周,在相同的条件下再用HIV病毒株JR-CSF感染细胞,继续培养10天,发现细胞活力或GFP表达没有变化,这表明剩余的细胞在敲除基因之后,对HIV感染具有抵抗力(图3)。
与亲本细胞系相比,大多数存活的突变体缺乏CD4或CCR5(图3),存活细胞群体在细胞表面保持CD4和CCR5的高表达,这表明筛选确定了HIV感染额外的宿主因子。再过3周,用FACS分离出有活力的GFP阴性和阳性细胞,并对GFP阴性细胞群、HIV感染前收集的初始细胞群和未感染HIV病毒繁殖6周细胞群进行测序,分析sgRNA的丰度差异,最后筛选出5个丰度变化最大的基因(图4),而且几乎所有靶向CXCR4(编码一种不被JR-CSF使用的HIV共受体)或编码GTP酶的RAP2A这两个对照基因的gRNA都没有被富集(图5)。
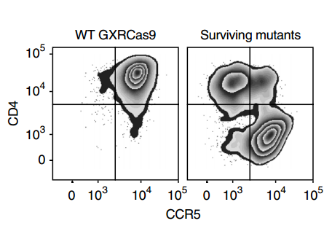
图3 流式细胞分析(代表3个独立实验)量化野生型GXRCas9细胞和用全基因组sgRNA文库转染并连续感染HIV-1株JR-CSF的GXRCas9细胞表面CD4和CCR5的表达。
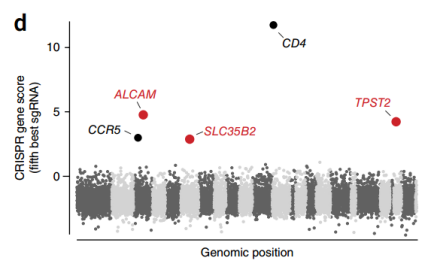
图4 HIV感染后对各基因的第5大富集sgRNA的丰度进行log2转化的折叠变化。
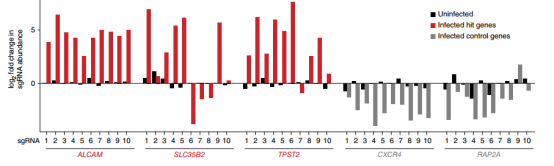
图5 三个候选宿主依赖因子和两个对照基因的单个sgRNA富集
为了探究TPST2、SLC35B2和ALCAM基因的缺失是如何抵抗HIV感染这一问题,作者使用CRISPR-Cas9系统构建了敲除这些基因的克隆GXRCas9细胞系并开展了一系列实验进行验证,结果表明,TPST2和SLC35B2可以促进HIV包膜识别CCR5,而ALCAM分子介导细胞间HIV传播,这一结果为治疗HIV病毒感染提供了潜在靶点。
参考文献:
[1] Park R J, Wang T, Koundakjian D, et al. A genome-wide CRISPR screen identifies a restricted set of HIV host dependency factors[J]. Nature genetics, 2017, 49(2): 193-203.










