总是筛不到纯合子克隆?你可能敲掉了必需基因!

总是筛不到KO纯合克隆?你可能敲除了必需基因!
引言
基因编辑经验丰富如你,是否遇到过以下诡异的场景:
1)细胞转染效率非常高:满屏都是blingbling的绿色荧光;
2)gRNA切割效率特别好:pool测序后PAM处的套峰肉眼可见;
可是单克隆鉴定结果一出来,要么是野生型(WT),要么是杂合子(KO+WT),要么是有敲除3的倍数基因型,重新来一次还是相同的结果…… 到底是怎么回事呢?
将目的基因从细胞中敲除,并观察由此产生的表型,是生物学研究中确定基因功能的常用策略,如用于研究细胞信号转导、基因表达调控或结构组成等。然而,有些基因对细胞活力至关重要,基因敲除后会导致细胞死亡,因此通过常规方法难以获得ko纯合克隆。这类基因被称为细胞生长的必需基因,引言中描述的现象很可能就是敲除了必需基因导致筛不到纯合子。
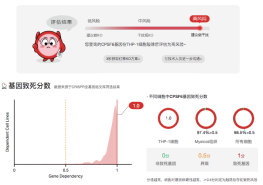
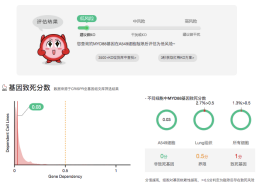
根据全基因组筛查结果表明,必需基因约占人类总基因的10%[1]。如果你不确定基因是否为细胞的必需基因,如何在实验前进行有效甄别,少走弯路呢?最先想到的方法可能是查找文献,看是否有相同细胞和基因敲除的成功案例。但是查文献毕竟费时间和精力,而且经常找不到完全一样的基因和细胞案例。随着CRISPR文库筛选的广泛应用,越来越多的细胞全基因组敲除数据被公开。源井生物汇集了1400+细胞系近3000w的海量敲除数据,只需输入细胞和基因名,即可一键评估项目致死风险(图1.2)并查找成功案例(图3)。点击免费使用>>>
高风险
| 低风险
|
| |
话说回来,如果恰好遇上高风险基因的敲除,只能束手无策吗?当然不是,源井生物根据多年细胞基因编辑经验,为你整理了3大解决方案。
1)退而求其次。如果基因彻底敲除后会导致细胞死亡,可以通过RNAi降低基因的表达观察是否有表型变化。如果基因敲低有表型,发文一般没问题。
RNAi文献案例:通过RNAi方法下调 MTA1的表达,可以导致ER α在ER阴性乳腺癌细胞系 MDA-MB-231中重新表达,并降低 MMP-9 和 CyclinD1 的蛋白水平,以及减少肿瘤细胞侵袭和增殖,更多的细胞被阻断在G0/G1期(P < 0.05)。MTA1的沉默作用可有效抑制MDA-MB-231细胞的侵袭和增殖。针对MTA1的shRNA干扰可能在人类乳腺癌中具有潜在的治疗效用[2]。
2)迎难而上,挑战概率。如果干扰效果确实不好,而基因也只是有一定致死概率的,可以尝试多筛一些克隆看能否幸运获得纯合子。如果没有获得纯合子,KO杂合子或者ko-pool也可以检测一下表达情况,有时候KO杂合子也有表型,可用于发文。
KO杂合子文献案例:NAT10 是一种核仁蛋白,包含一个乙酰转移酶结构域和一个tRNA结合结构域,具有组蛋白乙酰化活性,参与人端粒酶逆转录酶的调控。该基因在绝大部分肿瘤细胞中是必需基因,完全敲除后会导致细胞出现死亡。作者使用CRISPR技术在Lovo细胞中获得NAT10+/-的敲除杂合,进行免疫荧光、Western blotting和测序验证。与对照细胞 (LoVo) 相比,NAT10+/−细胞显示MN形成减少,即NAT10表达下调可以抑制结直肠癌进展[3]。
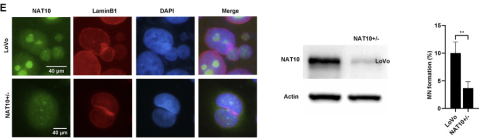
图4. NAT10 的表达下调减少了 MN 的形成
这里补充一点,有些敲除致死的基因,可以通过加入特殊添加剂,人为补充细胞生长所需养分,使细胞正常生长增殖,等到细胞扩增至足够量之后,再撤销添加剂,进行功能验证实验。比如白血病细胞系中敲除DHODH基因后,需要在培养基中添加~100µM的尿苷维持细胞生长[4]。
3)调整方案,条件性敲除。如通过使用tet-off系统和CRISPR/Cas9结合的方法,先用tet-off系统外源表达目的基因,再将内源基因敲除,筛选到KO纯合克隆扩增后,通过加Dox诱导外源基因表达关闭,实现基因诱导敲除[5]。或通过使用生长素诱导降解系统(Auxin-inducible degron,AID),将AID敲入形成融合蛋白替代内源基因表达,通过添加生长素进行蛋白诱导降解,实现基因功能敲除[6]。此外,也有将CRISPR/Cas9和cre/loxp系统结合,进行条件性敲除的策略。
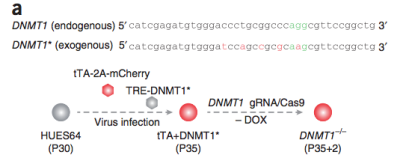
图5.条件性敲除策略1
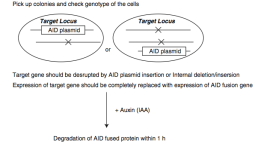
图6.条件性敲除策略2
以上3种解决方案,各有优缺点,第1种RNAi替代法操作最简单,成本较低,成功率高,但有可能出现干扰效果不好,敲除不彻底的问题。第2种加大范围筛选法,只适合部分致死率较低的基因,不确定性较高,时间和费用成本也比较高。第3种条件性敲除策略费用最高,tet-off系统也可能存在部分背景表达的情况,KI的方法则难度较高。总的来说,可以结合时间、经费和具体基因进行方案选择。
源井生物基因敲除现货库扩至3500+KO细胞,涵盖26条信号通路1200+基因,27种疾病类型1500+基因及5大类药物靶点800+基因,低至¥8000,1周达。更有4周极速定制,全方位方案评估和红棉基因风险分析,实验少走弯路,项目更有保障!
[1]Wang T, Birsoy K, Hughes N W, et al. Identification and characterization of essential genes in the human genome[J]. Science, 2015, 350(6264): 1096-1101.
[2] Jiang Q, Zhang H, Zhang P. ShRNA-mediated gene silencing of MTA1 influenced on protein expression of ER alpha, MMP-9, CyclinD1 and invasiveness, proliferation in breast cancer cell lines MDA-MB-231 and MCF-7 in vitro[J]. Journal of Experimental & Clinical Cancer Research, 2011, 30(1): 1-11.
[3] Cao Y, Yao M, Wu Y, et al. N-acetyltransferase 10 promotes micronuclei formation to activate the senescence-associated secretory phenotype machinery in colorectal cancer cells[J]. Translational Oncology, 2020, 13(8): 100783.
[4] Sykes D B. The emergence of dihydroorotate dehydrogenase (DHODH) as a therapeutic target in acute myeloid leukemia[J]. Expert opinion on therapeutic targets, 2018, 22(11): 893-898.
[5] Liao J, Karnik R, Gu H, et al. Targeted disruption of DNMT1, DNMT3A and DNMT3B in human embryonic stem cells[J]. Nature genetics, 2015, 47(5): 469-478.
[6] Nishimura K, Fukagawa T. An efficient method to generate conditional knockout cell lines for essential genes by combination of auxin-inducible degron tag and CRISPR/Cas9[J]. Chromosome Research, 2017, 25: 253-260.