CRISPR文库:猪病原性病毒研究

随着养殖技术的进步以及国内对猪肉需求量的不断增加,猪养殖业趋向于规模化方向发展,在部分地区,猪养殖业已成为当地重要的经济来源。然而,规模化养殖也带来了一些挑战,尤其是疾病防控问题。病毒性疾病由于其传染性强、死亡率高,对养殖业构成了严重威胁。因此,研究病毒与宿主之间的相互作用,了解病毒的传播机制和宿主的免疫反应,对于开发有效的预防和控制措施至关重要。
CRISPR文库筛选技术的发展,使得研究者们能够以低成本的方式,通过高通量、大规模筛选的方法,寻找新的抗病毒靶点。目前,针对常见的猪病毒,如口蹄疫病毒(FMDV)、经典猪瘟病毒(CSFV)、非洲猪瘟病毒(ASFV)以及猪流行性腹泻病毒(PEDV)等,已有使用CRISPR文库筛选出全新抗病毒靶点的相关研究,下面小源就挑选其中两篇为大家简单介绍一下吧。
TOB1是FMDV病毒感染的关键宿主因子
文库类型:猪全基因组敲除文库
转导细胞:IBRS-2
筛选思路:使用FMDV连续感染文库细胞两轮,并分别收集细胞用NGS测序。
筛选方法:使用0.2的MOI对文库细胞感染FMDV病毒,感染1.5h后,弃上清,换用含2%血清培养基培养细胞10天,收集部分细胞用于NGS测序,剩余细胞重复一轮感染(图1)。
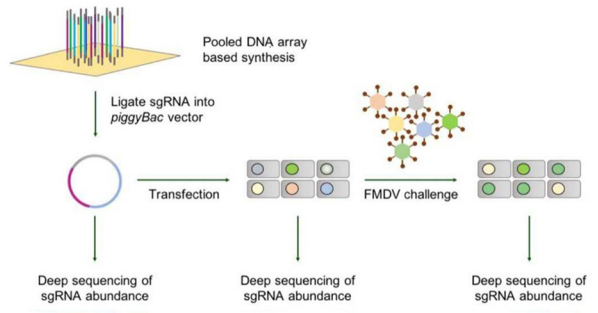
图1 FMDV感染筛选方法[1]
通过上述文库筛选实验,研究者们根据sgRNA的丰度对候选基因进行了识别和排序,并计算出了基因得分,随后对排名前30的基因在IBRS-2细胞中进行了敲除验证,发现其中7个基因敲除后能显著抑制FMDV的感染,并且TOB1基因敲除对病毒感染的抑制能力最强(图2)。
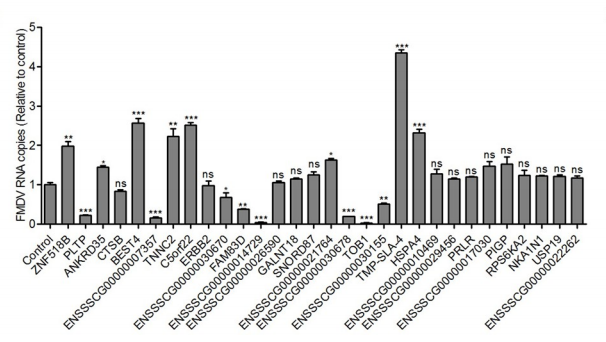
图2 基因敲除验证[1]
研究人员进一步对TOB1基因进行了下游验证:在3个不同的细胞系(IBRS-2、PK-15和iPAM)中对其进行基因敲除后,发现TOB1基因敲除后通过促进RIG-I 和MDA5的表达来激活IFN-β的表达(图3),并且通过抑制EGFR信号通路来抑制FMDV病毒的感染(图4)。
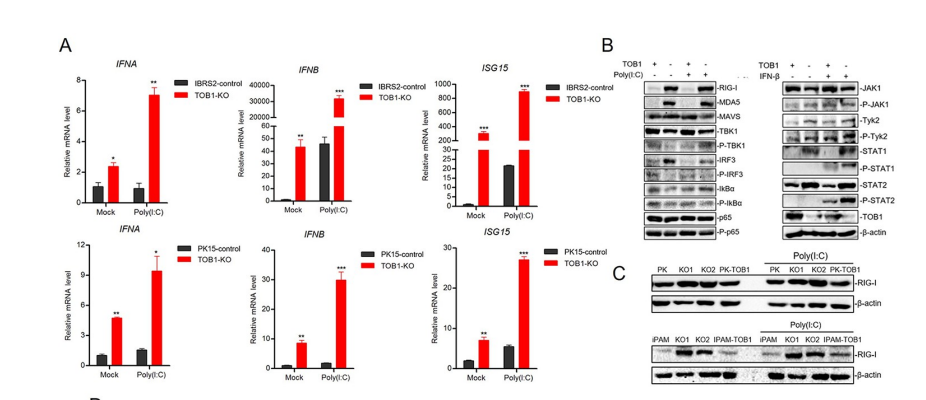
图3 TOB1基因敲除促进RIG-I 和MDA5的表达[1]
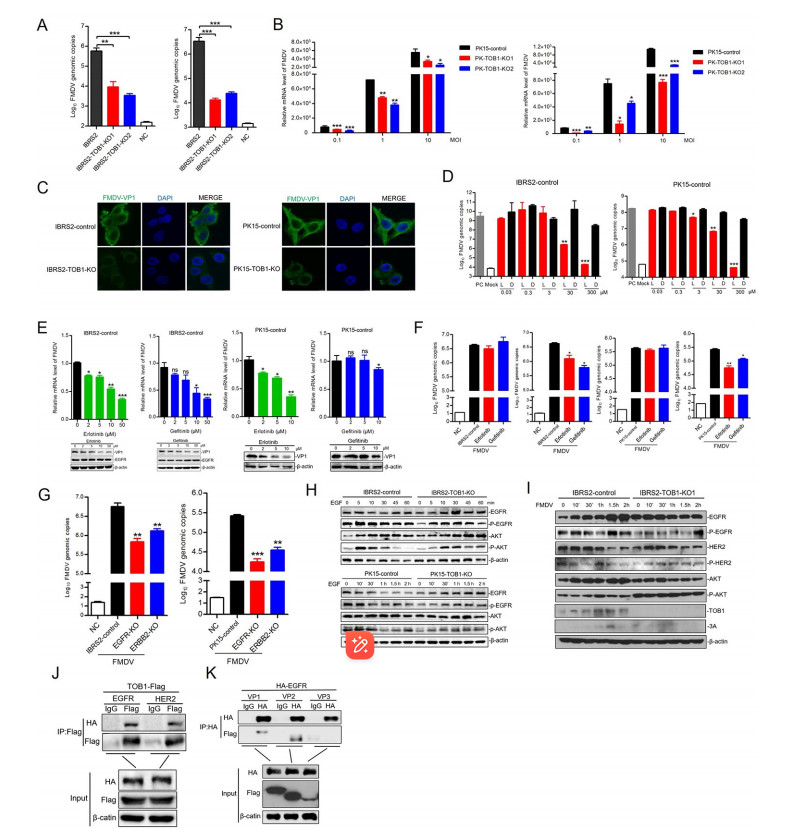
图4 TOB1基因敲除抑制EGFR信号通路表达[1]
TRIM2和SLC35A1与PEDV病毒感染相关
文库类型:猪全基因组敲除文库
转导细胞:Vero E6
筛选思路:使用不同病毒量分别感染细胞,并收集细胞用于NGS测序。
筛选方法:使用MOI为0.00001和0.0001的病毒量分别感染Vero E6细胞一周后,收取存活细胞用于NGS测序分析。
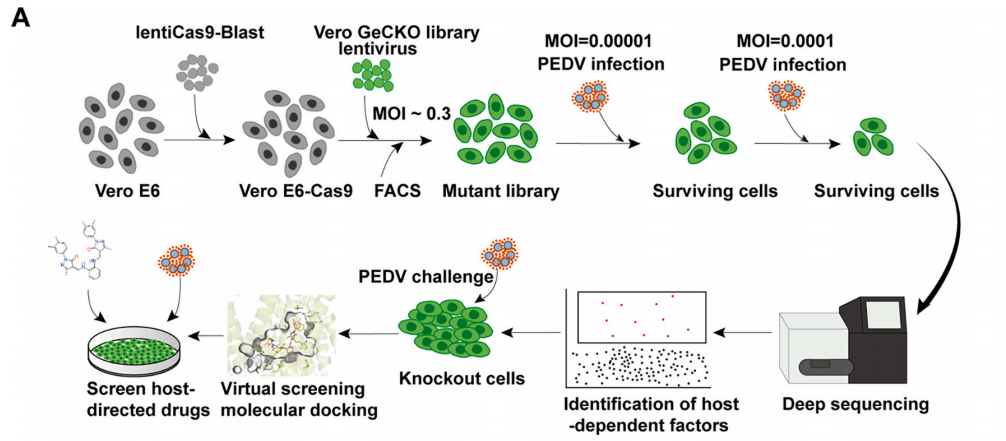
图5 PEDV病毒感染筛选流程[2]
由于绿猴细胞Vero E6对PEDV感染敏感,并被广泛用于PEDV病毒的分离和培养,研究人员选择使用该细胞株进行文库筛选。通过sgRNA的差异表达倍数对基因进行排序后,研究人员富集了sgRNA表达倍数差异排名前20的基因,并对其中8个进行了基因敲除验证,发现这些基因敲除均能显著抑制病毒感染后细胞的死亡,并且减少细胞中的病毒拷贝数(图6)。
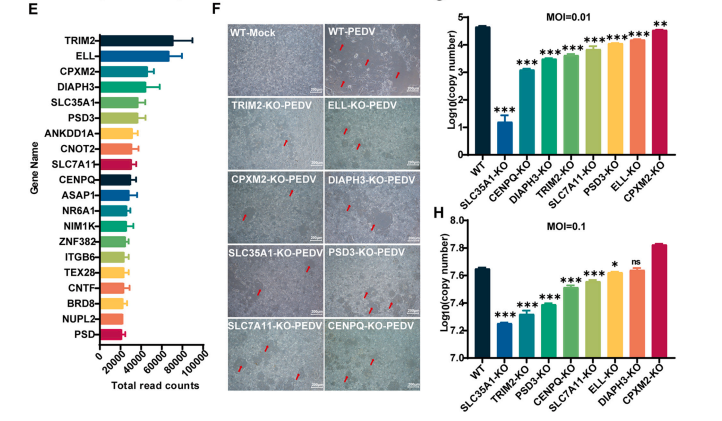
图6 各基因敲除细胞病毒感染验证[2]
通过qPCR检测病毒感染后TRIM2的表达,研究人员发现该基因的表达随着感染时间的延长而升高,结合免疫荧光检测细胞中PEDV病毒衣壳蛋白的表达,发现TRIM2基因敲除后细胞中PEDV病毒衣壳蛋白含量显著降低(图7),说明TRIM2对于PEDV病毒的感染十分重要。研究人员进一步检测SLC35A1基因敲除细胞系中PEDV病毒衣壳蛋白的表达并结合SLC35A1基因的小分子拮抗剂,发现SLC35A1基因敲除能减缓病毒感染后细胞的死亡(图8)。
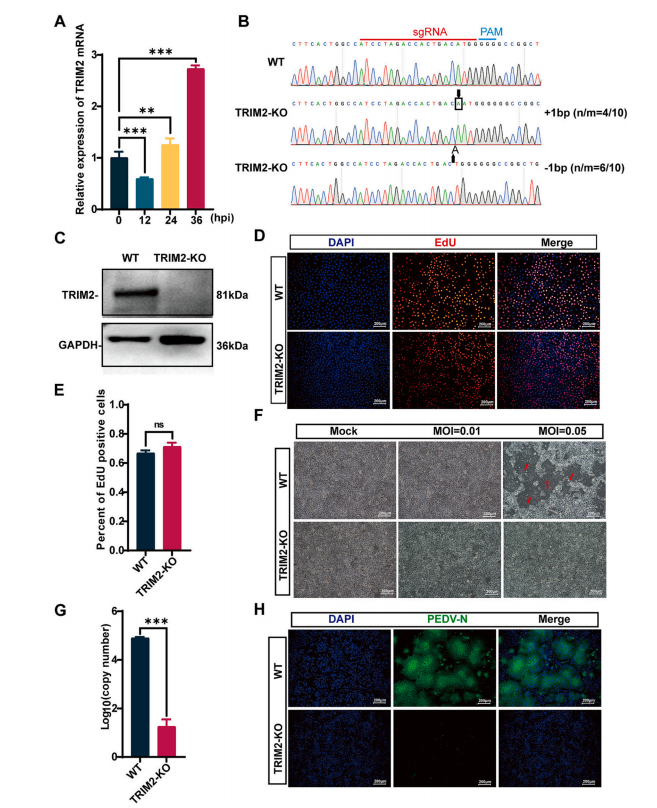
图7 TRIM2基因敲除显著抑制PEDV病毒感染[2]
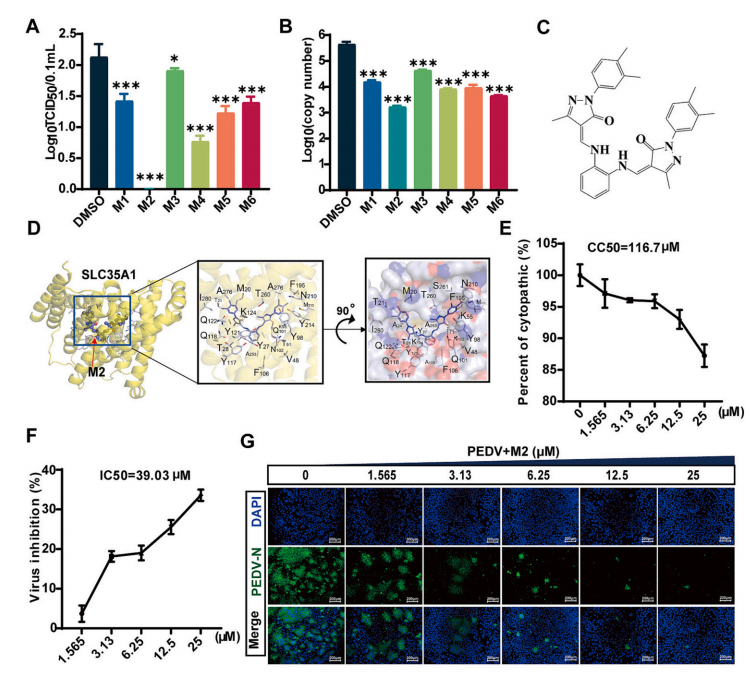
图8 SLC35A1拮抗剂抑制PEDV病毒感染后细胞死亡[2]
关于CRISPR文库用于猪病毒靶点筛选相关的研究远不止于此,如ASFV[3]、猪繁殖与呼吸综合征病毒(PRRSV)[4]和乙型脑炎病毒(JEV)[5]等也有相关报道。
各种病毒由于其致死性强、传染性高等特点,对养殖业造成了严重困扰,家畜类的抗病毒因子的筛选对于养殖业具有重大意义。基于CRISPR文库筛选技术在家畜类病毒的抗病毒靶点筛选研究中已被广泛应用,除了上述小源为大家介绍的用于猪病毒相关的研究外,在其他家畜,如鸡、狗、牛等相关研究中也常有报道。
参考文献
[1] Peng G, Liu T, Qi X, Wang Y, Ren J, Peng J, Du X, Hu S, Wu S, Zhao Y, Li D, Zheng H. A genome-wide CRISPR screening uncovers that TOB1 acts as a key host factor for FMDV infection via both IFN and EGFR mediated pathways. PLoS Pathog. 2024 Mar 21;20(3):e1012104.
[2] Wang J, Liu H, Yang Y, Tan Y, Sun L, Guo Z, Zeng X, Wang Z, Li S, Yin L, Yin D, Shen X, Dai Y, Liu X, Ruan J, Li X, Zhao S, Peng G, Pan X, Wang C, Xie S. Genome-scale CRISPR screen identifies TRIM2 and SLC35A1 associated with porcine epidemic diarrhoea virus infection. Int J Biol Macromol. 2023 Oct 1;250:125962.
[3] Pannhorst K, Carlson J, Hölper JE, Grey F, Baillie JK, Höper D, Wöhnke E, Franzke K, Karger A, Fuchs W, Mettenleiter TC. The non-classical major histocompatibility complex II protein SLA-DM is crucial for African swine fever virus replication. Sci Rep. 2023 Aug 21;13(1):10342.
[4] Jiang J, Sun Y, Wang Y, Sabek A, Shangguan A, Wang K, Zhao S, Li G, Zhou A, Zhang S. Genome-wide CRISPR/Cas9 screen identifies host factors important for porcine reproductive and respiratory syndrome virus replication. Virus Res. 2022 Jun;314:198738.
[5] Zhao C, Liu H, Xiao T, Wang Z, Nie X, Li X, Qian P, Qin L, Han X, Zhang J, Ruan J, Zhu M, Miao YL, Zuo B, Yang K, Xie S, Zhao S. CRISPR screening of porcine sgRNA library identifies host factors associated with Japanese encephalitis virus replication. Nat Commun. 2020 Oct 14;11(1):5178.
 CRISPR基因编辑技术服务
CRISPR基因编辑技术服务
 红棉 · 基因敲除计划
红棉 · 基因敲除计划


























