Nature| 源井KO细胞助力揭秘中国人群肝癌全基因组变异及演化特征

Nature| 源井KO细胞助力揭秘中国人群肝癌全基因组变异及演化特征

引言
原发性肝癌的致死率在全球所有癌症中高居第三,肝细胞癌(hepatocellular carcinoma, HCC)为其主要亚型,约占其所有病例的80%以上。全球每年肝癌新发病例超过一半发生在中国,且患者预后普遍较差,我国肝细胞癌患者五年总体生存率仅约为10%。肝细胞癌病因复杂,其中HCV感染在日本人群最为常见,酒精、非酒精性脂肪性肝炎及代谢紊乱在西方人群中占比较大,而HBV慢性感染和黄曲霉毒素等致癌物暴露是中国肝癌人群的主要致病因素。
既往的国际泛癌全基因组研究计划(Pan-Cancer Analysis of Whole Genomes, PCAWG)和美国癌症基因组研究计划(The Cancer Genome Atlas, TCGA)虽然包含了对肝癌全基因组特征的研究,但其样本主要为日本和西方人群,其结果不能全面反映中国人群肝癌的基因组特征。另一方面,这些研究采用了相对较浅的测序深度(~30-40X),限制了其对全基因组层面亚克隆结构的深度解析。鉴于不同区域、不同人群肝癌致病因素的巨大差异,迫切需要开展一项大规模、高深度的中国肝癌人群全基因特征分析,为开发适用于中国肝癌人群的诊断和治疗方案提供依据。
摘要:
2024年2月14日,海军军医大学王红阳院士/陈磊研究员课题组联合北京大学生物医学前沿创新中心/北京未来基因诊断高精尖创新中心白凡教授课题组、杜克-新加坡国立大学医学院Steven G. Rozen教授课题组以及福建和瑞基因吴琳博士团队合作在Nature在线发表了题为 Deep whole-genome analysis of 494 hepatocellular carcinomas 的最新研究成果,完成了中国人群肝细胞癌全基因组深度特征分析(Chinese Liver Cancer Atlas, CLCA)。本研究对494例来自中国不同地区的肝细胞癌患者肿瘤组织进行了高深度的全基因组测序(平均120x),深入分析了编码区和非编码区的驱动基因、突变印记、拷贝数变异、聚集式变异事件(clustered alteration:chromothripsis, chromoplexy和kataegis)、染色体外环状DNA(extrachromosomal circular DNA, ecDNA)以及突变演进规律等特征。还选取了3个新鉴定的潜在驱动事件进行详细功能验证,综合CRISPR定点突变及多个细胞系敲降/敲除实验,发现上述基因的突变足以导致基因表达水平的显著变化(其中PPP1R12B, KCNJ12, FGA基因敲除细胞和携带突变位点的过表达慢病毒均由源井生物构建),并参与调控肝细胞癌的多种恶性表型,这些结果证实基于数据分析发现的新驱动事件的有效性。

研究方法及结果
先前对中国个体HCC的基因组分析仅限于队列大小,并且主要关注外显子组,排除了全基因组水平的详细研究。最近,泛癌症全基因组分析(PCAWG)联盟在相当大的范围内分析了癌症的基因组复杂性。然而,相对较浅的测序深度并不能完全解析HCC基因组的亚克隆结构。在CLCA中,研究人员对494个HCC肿瘤(平均深度,120×)以及匹配的对照血液样本(平均深度,36×)进行了深度全基因组测序(WGS)分析。队列包括427名男性(86.4%)和67名女性(13.6%)。与PCAWG-HCC (n = 248)队列相比,CLCA队列的HBV感染比例(94.5%对30.6%)和edmonson - steiner分级3级和4级(85.6%对12.1%)较高,但丙型肝炎病毒(HCV)感染比例(2.6%对55.6%)、饮酒(26.7%对58.1%)和吸烟(36.8%对53.6%)较低。这些统计数据代表了中国肝癌人群的流行病学,突出了当前研究的必要性。
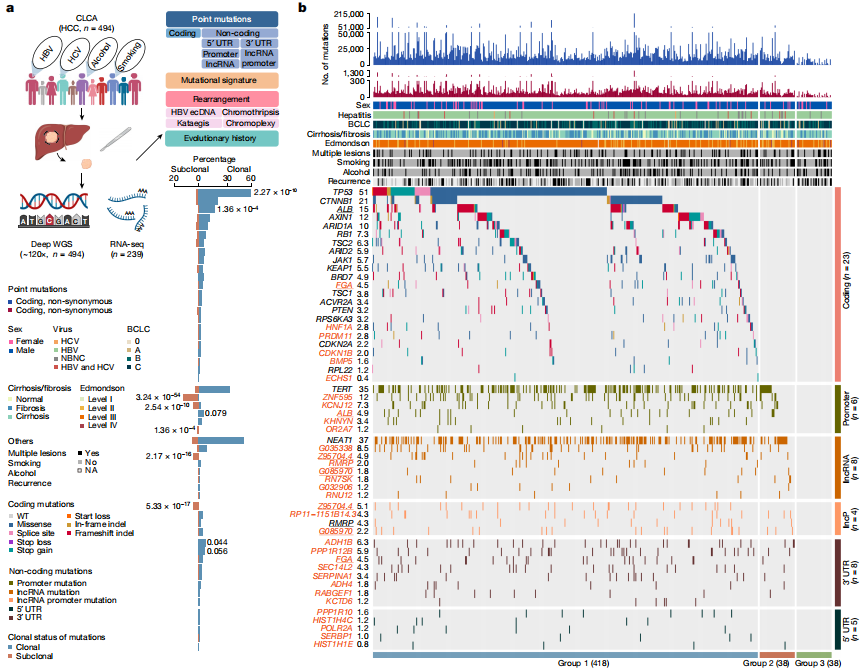
图1.候选驱动基因景观
经过严格的质量控制,共鉴定出9,287,828个体细胞突变,每个肿瘤中位数为13,735.5个突变和95个非同义突变。研究人员还对来自该队列的239个肿瘤进行了RNA测序(RNA-seq)分析。该研究确定了6个编码和28个非编码先前未描述的驱动候选基因。该研究发现了五个先前描述的突变特征,包括马兜铃酸相关的indel和双碱基特征,以及命名为SBS_H8的单碱基取代特征。五核苷(Pentanucleotide)酸背景分析和实验验证证实,SBS_H8与马兜铃酸相关的SBS22是不同的。
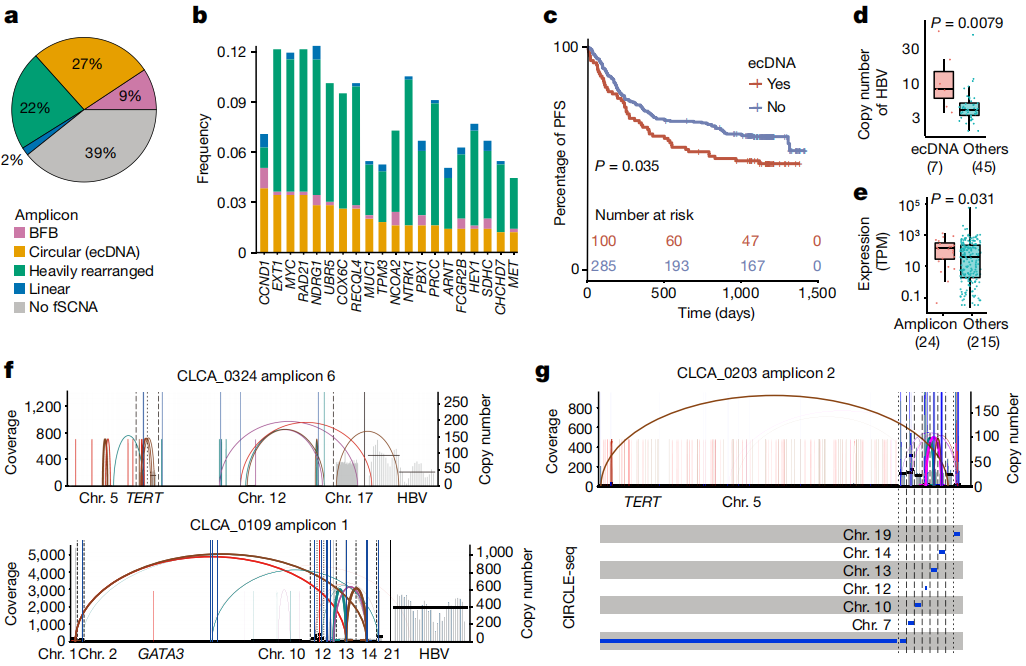
图2 ecDNA分析
值得注意的是,HBV整合可能采取染色体外环状DNA的形式,导致拷贝数和基因表达升高。高深度数据还使研究人员能够表征亚克隆群集性改变,包括染色体碎裂(chromothripsis)、染色体重排(chromoplexy)和基因组区域发生的多个突变簇(kataegis),这表明这些灾难性事件也可能发生在肝癌发生的晚期。所有类型改变的通路分析进一步将非编码突变与肝脏代谢失调联系起来。最后,该研究进行了体外和体内实验,证明纤维蛋白原α链(FGA)作为候选编码和非编码驱动因子,调节HCC的进展和转移。
结论
综上,本研究所建立的中国肝癌人群基因组变异特征全景图谱,为深入理解不同病因肝癌的发生和演进分子机制、开发全新的个体化靶向药物提供了宝贵资源,有助于提升中国肝癌的临床精准诊疗水平,造福广大肝癌患者。










