一招超快研究基因功能--基因敲除细胞混合克隆(KO pool)
CRISPR是一种革命性的基因组编辑技术,是一项有可能会改变世界的技术。虽然这还是一个相对较新的发现,但很快,CRISPR将成为用于生物学研究标准的实验工具。CRISPR最广泛应用之一是敲除特定基因以了解该基因的功能。实验通常需要同时转染大量细胞,形成具有不同Indel的基因编辑细胞混合克隆,也被称为KO(敲除)pool。CRISPR介导产生的KO pool取代了较传统的基于RNAi的方法,正逐渐成为许多研究细胞系基因功能的研究人员的首选方法。KO pool混合了野生型和编辑过的细胞,敲除效率可能会随时间而变化。但是,对于短期测定来说,KO pool是非常有用的。
具有高敲除效率的KO pool适合直接用于许多类型的测定,而不需要额外挑取单克隆细胞来进行测定,因为基因型畸变可以直接影响到表型状态。因此,KO pool在以下研究领域具有广泛的应用:
1. 增殖测定
2. 基因发现:识别新的癌症驱动基因并发现癌症特异性脆弱性。
3. 药物反应/药物靶标识别
4. 疾病模型发展
5. 抗体验证(免疫印迹/免疫荧光)
CRISPR/Cas9系统是一种有效的基因组编辑系统,通过用gRNA和所需的donor序列打靶和取代靶基因序列,进行基因敲除或敲入的基因组编辑操作。通过CRISPR技术制作KO pool,可节省单克隆化而花费的时间和成本。如今,科学家开始从RNAi转移到使用KO pool进行基因的早期探索性研究,因为KO pool降低了进入的成本,并且与RNAi非常相似。此外,CRISPR技术可以产生稳定的蛋白质功能敲除,可以提供比RNAi更彻底的结果。具有更高效率的KO pool对于药物靶标,药物反应,抗体验证,疾病模型开发以及各种功能测定的研究来说非常有用。为了加快生物医学研究的发展,源井生物开发了CRISPR-U™系统,应用于快速而精确的细胞基因编辑。此外,CRISPR产生的KO pool可以与功能丧失测定,高通量筛选,抗体验证和安全药理学研究相结合。源井生物可以定制基因编辑敲除细胞系和KO pool。

图: 定制CRISPR-U™ KO pool的工作流程
源井生物开发的CRISPR-U™优化细胞的基因编辑。其效率和准确性高于传统方法。请立即联系我们了解与您的研究相关的服务吧!
KO pool可用于研究表达量过低或不可检测的靶蛋白基因
基于CRISPR/Cas9的基因敲除(KO)能够精确扰乱靶基因的功能,并且在理想情况下,通过人类细胞中的分子组学读数将以无偏差的方式分析这些现象。一般来说,这需要一个分离KO单克隆的漫长过程。在这项研究中,研究人员使用了一种策略,避免了进行单克隆分选,并通过使用在基因组上打靶接近序列(40-300 bp)的两个gRNAs的协同组合,在KO pool上实现了快速而有效的基因沉默。研究人员在HepG2细胞中敲除吡哆醛激酶(PDXK)并生成PDXK-KO pool。同时,研究人员还制备PDXK-KO单克隆来进行比较。他们比较了KO单克隆和KO pool中产生的表型蛋白质组学数据。
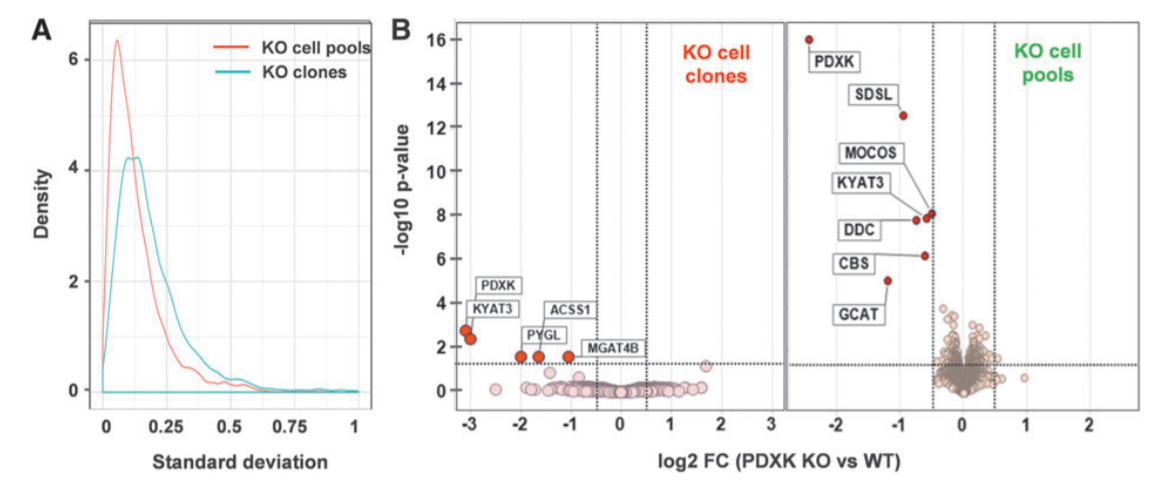
图1: PDXK-KO细胞的蛋白质组学表型
重复实验之间的标准差显示了KO pool的数据集比三个PDXK-KO单克隆数据集显示出更小的变化(见图1A)。无偏差全蛋白质组分析在HepG2 KO pool中鉴定出了6种显着下调的蛋白质,而在KO克隆中,只鉴定出4种显着下调的蛋白质。我们需要用MS定量对同源蛋白的残留水平进行检测,以排除该截短蛋白在早期终止密码子或外显子跳跃后仍表达,而这可能会扰乱KO表型的分析和解释。
因此,基因敲除(KO)pool同样能够精确地干扰靶基因功能,可通过分子组学读数评估基因敲降以及分析KO表型。使用KO pool,可能是比RNAi更好的研究基因功能的方法。
源井生物开发的CRISPR-U™优化了真核细胞和动物基因编辑载体和过程。效率和准确度比传统方法高10倍。立即联系我们了解与您研究相关的基因编辑服务!
Reference::
A Tandem Guide RNA-Based Strategy for Efficient CRISPR Gene Editing of Cell Populations with Low Heterogeneity of Edited Alleles. CRISPR J.2020;3(2):123-134.
 CRISPR基因编辑技术服务
CRISPR基因编辑技术服务
 红棉 · 基因敲除计划
红棉 · 基因敲除计划



























