CRISPR/Cas9构建稳定敲除细胞系的实用指南—— 从 sgRNA 设计到单克隆验证的系统流程

CRISPR/Cas9构建稳定敲除细胞系的实用指南—— 从 sgRNA 设计到单克隆验证的系统流程
引言
CRISPR/Cas9 基因编辑技术以其高效、简便和可编程性,已成为研究基因功能、疾病机制及药物靶点验证的核心工具。相比瞬时编辑,稳定敲除细胞系(Stable Knockout Cell Line) 能在多代传代中保持一致的基因缺失状态,确保实验结果的重复性与可靠性,是基础研究与药物开发中不可或缺的模型系统。然而,稳定敲除细胞系的构建是一个多环节、系统性工程,涵盖了从 sgRNA 设计到单克隆筛选的多个关键步骤。小源将系统介绍利用 CRISPR/Cas9 构建稳定敲除细胞系的标准流程,并说明如何借助源井生物(Ubigene)的专业技术平台,实现更高效、更精准的基因编辑结果。
一、 实验前准备
在实验开始前,应全面评估目标基因及细胞特性,确保设计方案科学合理。主要包括以下环节:
- · 基因信息确认:检查目标基因的转录本差异、可变剪接情况和功能结构域(可参考 Ensembl、NCBI 等数据库)。
- · 细胞特性评估:了解细胞的转染效率、生长速率、药物敏感性及是否属于难转染类型(如原代细胞、干细胞、免疫细胞等)。
- · 基因可编辑性评估:参考 DepMap、Gene Essentiality 等公开数据库,或者使用源井生物的Gene Dependency Assessment系统,判断基因是否为必需基因,避免因敲除导致细胞死亡或增殖障碍。
- · 下游验证方法准备:提前规划验证方案,包括蛋白检测(Western Blot)、qPCR、功能实验以及一代测序(Sanger Sequencing)等。
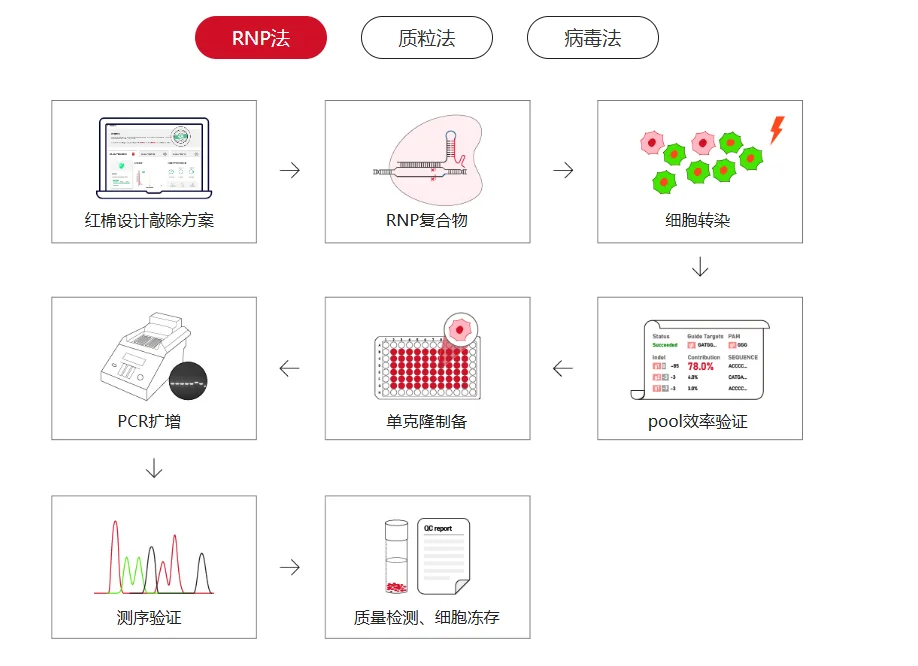
图一:敲除细胞系构建流程
二、 实验步骤
1. sgRNA 与基因型鉴定引物(Genotyping Primer)的设计与合成
- · sgRNA 设计:通过分析目标基因序列,选择合适的靶点。建议优先选择靶向关键功能结构域外显子的 sgRNA,以实现更彻底的基因失活。设计时应综合考虑以下因素
- · On-target 编辑效率评分
- · 潜在脱靶效应(可使用 CRISPOR、Benchling 、红棉CRISPR基因编辑系统等工具)
- · 外显子位置、阅读框影响及 PAM 序列可及性
- · 引物设计:根据 sgRNA 靶点上下游序列设计 PCR 引物,扩增长度建议控制在 250–800 bp 之间,以便后续突变检测(T7E1 断裂实验或 Sanger 测序)。
2. 载体构建
将 sgRNA 序列克隆至含有 Cas9 蛋白及筛选标记(如嘌呤霉素、EGFP)的载体中。源井生物的红棉 gRNA 载体库拥有超过 10,000 种现货载体,采用优化的载体骨架结构,可显著提升 sgRNA 表达与 Cas9 切割效率,特别适用于难转染细胞及多靶点联合编辑研究。 可广泛应用于肿瘤学、神经科学、代谢疾病及细胞治疗等研究领域。
3. 质粒验证
通过测序验证 sgRNA 插入的准确性与序列完整性,确保无突变、插入缺失或方向错误。质粒验证是整个实验的质量控制关键,可有效避免假阴性或非特异编辑结果。
4. 质粒制备与质量检测
制备高纯度、无内毒素质粒,用于细胞转染。检测 A260/A280 比值(应为 1.8–2.0),并使用琼脂糖电泳确认质粒完整性。内毒素会影响转染后的细胞生理状态,低纯度DNA含有各种杂质影响转染效率。
5. 转染前优化
为确保编辑效率,建议进行充分的准备工作:细胞处于对数生长期、无支原体污染、细胞密度适宜(一般为 70–90% 融合度)、根据细胞类型选择最佳转染方式(化学法、电穿孔、病毒转染等)。
6. 细胞转染
将 CRISPR/Cas9-sgRNA 质粒导入细胞。设置适当对照:空载体阴性对照、阳性对照(编辑已知可见表型基因)、通过荧光或抗性筛选监测转染效率。
7. Cell Pool 筛选与鉴定
转染 48–72 小时后收集细胞群体(Cell Pool),提取基因组 DNA 进行突变检测。常用检测方法包括:
- · PCR + Sanger 测序:精确鉴定插入/缺失类型(indel)
- · T7E1 或 Surveyor 断裂实验:评估编辑效率与突变比例。
源井生物采用 PCR + 测序双重检测法评估编辑效率,并通过并行策略筛选最佳敲除效率的细胞群,为后续单克隆筛选节省时间。源井生物拥有Genotype Analysis System可以一键拆解测序结果,并完成分析,可快速获取Cell Pool的敲除效率结果。
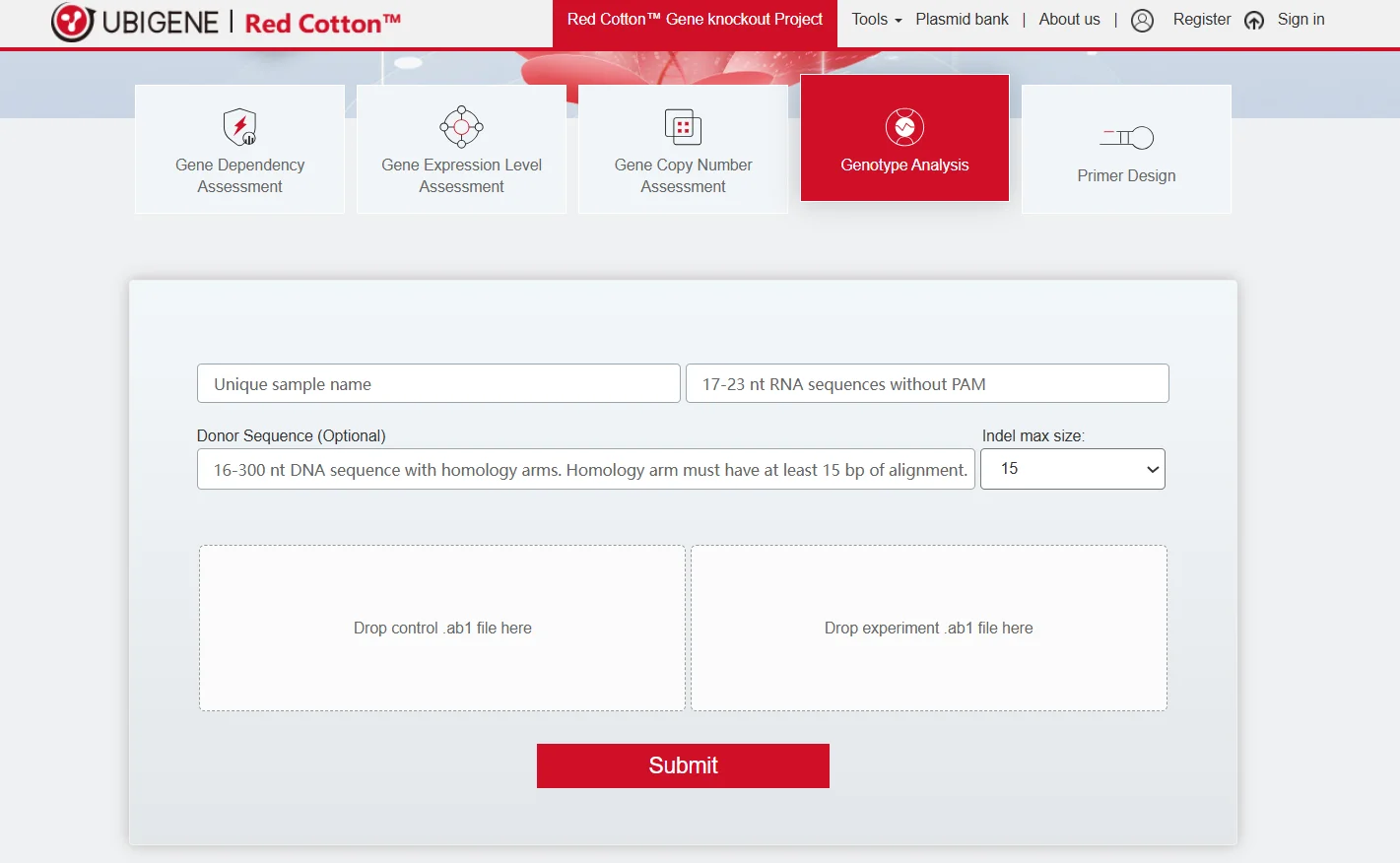
图二:源井生物基因型分析系统
8. 单克隆细胞筛选与扩增
通过有限稀释法或流式分选(FACS)将细胞分配至单孔,实现单细胞克隆培养。扩增后提取基因组 DNA,再次进行突变检测,确认敲除类型(Homozygous/bi-allelic KO)。经验证的单克隆应进行功能验证,如蛋白表达丢失或信号通路变化,以确保真正实现基因敲除。
9. 质量检测与冻存
稳定敲除细胞系建立后,应进行以下质量控制步骤
- · 支原体检测:确保无污染
- · STR 鉴定:验证细胞身份一致性
- · 功能验证:检测靶基因表达及相关表型
通过质量检测的敲除细胞系应及时冻存(如使用含 10% DMSO 的 FBS 冻存液),以备后续使用。
三、 源井生物敲除细胞系服务推荐
构建稳定敲除细胞系过程复杂,对技术经验与细胞特性要求较高。源井生物(Ubigene) 提供从设计到交付的一站式基因敲除解决方案,帮助科研人员高效获得可靠的模型系统。源井生物的核心优势包括:
- · CRISPR-U™ 技术平台:编辑效率提升 10–20 倍,复杂细胞成功率达 90% 以上
- · 红棉CRISPR基因编辑系统: 基于 AI 算法与1000+ 细胞参数数据库,实现自动化设计与风险评估;
- · 8000+ 标准化敲除细胞库:提供即用型细胞模型,满足多领域研究需求;
- · 全流程质控体系:STR 鉴定、支原体检测、功能验证,确保细胞质量稳定可靠。
凭借累计 8000+ 成功项目经验,源井生物为全球科研机构和药企提供高质量敲除细胞系,显著缩短研发周期。
四、 结语
CRISPR/Cas9 构建稳定敲除细胞系是解析基因功能与疾病机制的关键手段。通过科学的 sgRNA 设计、优化的转染策略以及严格的筛选验证,可获得高质量、可重复的敲除模型。若希望在保持实验可控性的同时,提升效率与成功率,源井生物的专业敲除细胞系服务可为科研人员提供高效、可靠、省时的整体解决方案,助力生命科学研究与创新。











