筛靶点:CRISPR文库vs传统组学?

筛靶点:CRISPR文库vs传统组学?

在生命科学研究和精准医学领域,靶点筛选是一项至关重要的任务。它为疾病的诊断、治疗和药物研发提供了关键的方向。CRISPR技术的出现无疑为这一领域带来了革命性的变化。靶点筛选方法多种多样,通常应用传统组学方法和CRISPR文库筛选方法,但每种方法都有其优势和局限性,一起来了解一下吧。
传统组学方法筛靶点
传统组学方法在药物靶点筛选中扮演着重要角色,主要包括基因组学、转录组学、蛋白质组学和代谢组学等。这些方法通过对生物样本中基因、转录本、蛋白质或代谢物的大规模检测与分析,揭示生物系统在不同生理或病理状态下的变化规律,从而找出可能与疾病相关的靶点。
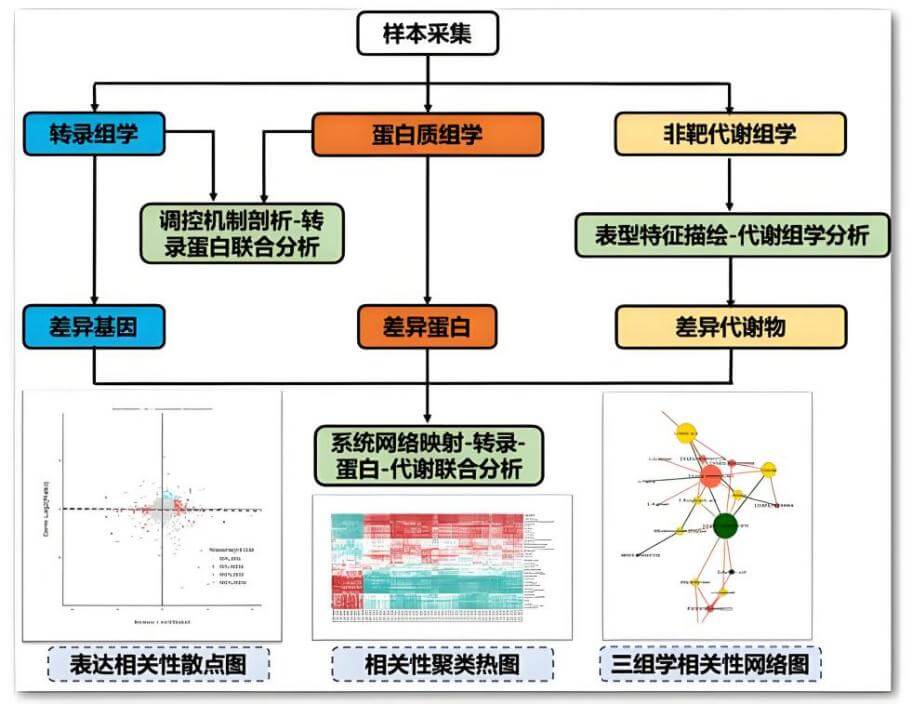
图1.传统组学筛靶点
基因组学(Genomics):侧重于研究生物体的全部基因组信息。例如,全基因组关联分析(GWAS)通过比较大量病例与对照人群的基因组序列,寻找与疾病性状显著关联的单核苷酸多态性(SNP)位点。这些关联位点可能位于基因的编码区或调控区域,从而影响基因的功能或表达水平,进而与疾病的发生发展相关联。
转录组学(Transcriptomics):主要研究细胞在特定条件下转录出来的所有RNA转录本的集合。通过RNA测序(RNA-seq)技术,可以获取基因的表达水平信息,分析疾病组织与正常组织之间差异表达的基因。
蛋白质组学(Proteomics):研究细胞或组织中的蛋白质组成、表达水平、修饰状态以及蛋白质之间的相互作用。基于质谱技术的蛋白质组学分析能够鉴定大量蛋白质,并定量分析其在不同样本中的差异。
代谢组学(Metabolomics):研究生物体内代谢物的种类、数量及其变化规律。通过对血液、尿液、组织等样本中的代谢物进行检测,如气相色谱-质谱联用(GC-MS)、液相色谱-质谱联用(LC-MS)等技术,可以发现疾病状态下代谢途径的紊乱。
CRISPR文库筛选靶点
CRISPR/Cas9文库筛选技术是一种高效的基因功能研究方法。通过构建包含大量针对不同基因的导向RNA(gRNA)文库,并将其导入细胞群体中,利用Cas9蛋白对基因组进行编辑。然后通过特定的筛选策略,如细胞存活、增殖、表型变化等,可鉴定出与特定生物学过程或疾病相关的基因。
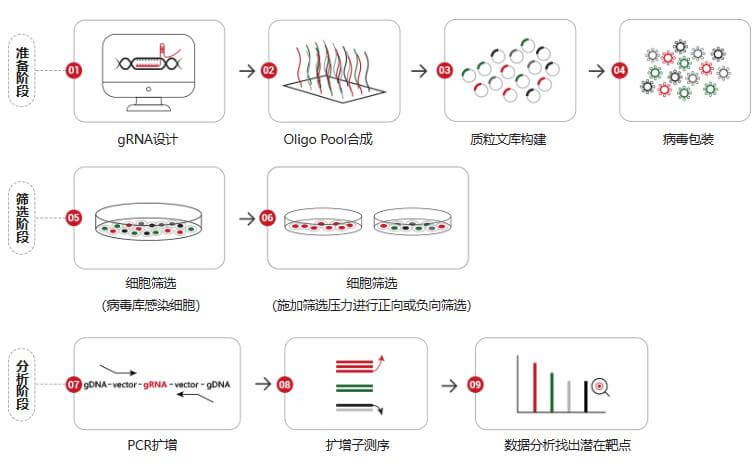
图2.CRISPR文库筛选靶点流程
CRISPR文库筛选与传统组学方法筛靶点对比
| 对比 | CRISPR文库筛选 | 传统组学方法 |
| 优势 | 1. 直接评估基因功能 2.高通量功能验证 3.可发现新的药物靶点 4.快速验证因果关系 | 1.可以获得系统性的分子图谱; 2.适用于临床样本分析; 3.能够发现新的分子亚型; 4.有助于理解疾病异质性 |
| 局限 | 1. 主要局限于体外实验 2. 可能存在脱靶效应 3. 文库设计和质量控制要求高 4. 某些基因可能难以编辑 | 1. 相关性分析难以确定因果关系数据分析复杂, 2. 需要强大的生物信息学支持 3. 功能验证周期长 |
CRISPR文库优势
1. 功能验证方面
CRISPR文库筛选技术可直接研究基因的功能缺失或功能获得,明确基因在特定生物学过程中的因果关系。
2.筛选全面性
CRISPR文库筛选可在全基因组范围内无偏向性地筛选,理论上能够发现任何与筛选条件相关的基因,包括一些尚未被研究或功能未知的基因。
3.靶点可操作性
相比于传统组学方法筛选出蛋白质、代谢物等靶点,后续需复杂的药物设计研发过程(如开发蛋白质靶点小分子抑制剂或抗体药物),CRISPR文库筛选鉴定的靶点多为可直接编辑的基因,便于后续基因治疗药物研发,如单基因遗传病致病基因,可直接开发基因编辑策略。
应用实例:可以点击查看往期文章,文库应用系列 | CRISPR文库筛选在急性白血病研究中的应用,CRISPR文库丨解锁乳腺癌靶点筛选新思路!,CRISPR文库:为病毒研究打开新视角——初阶干货(一)等等。除此,还有很多CRISPR文库干货,欢迎查看,后续我们也将上线更多CRISPR文库相关的内容,敬请期待~
基于以上优势,越来越多科研er选择CRISPR文库筛选新靶点,发高分文章!但是CRISPR文库筛选技术复杂,需构建高质量文库、精确细胞转导及高通量测序分析,依赖专业实验室与人员。
源井生物CRISPR-iScreen™平台,可提供从高通量sgRNA文库构建到病毒包装、细胞转染、药物筛选、高通量测序和数据分析等一站式服务。源井CRISPR-iScreen™技术,使用自主研发高效感受态及独家Cell Pool制备工艺,确保Cell Pool文库覆盖度高达99%,还拥有400+CRISPR文库现货产品,欢迎咨询~

参考文献:
[1] Joung JK, et al. Genome - scale CRISPR - Cas9 knockout and transcriptional activation screening. Nat Protoc. 2017;12 (4):828 - 863.
[2] Doench JG, et al. Genome - wide CRISPR - Cas9 knockout screening in human cells. Science. 2016;353 (6304):aaf8729.
[3] Luo B, et al. Genome - wide and pathway - focused CRISPR - Cas9 screens identify mediators of cancer drug resistance. Cell. 2015;161 (6):1247 - 1261.
[4] Reuveni E, et al. Integrating CRISPR screens with multi - omics data to decipher gene regulatory networks. Nat Rev Genet. 2017;18 (7):477 - 490.
[5] Tzelepis K, et al. CRISPR - Cas9 screens in cancer cells to identify novel drug targets and resistance mechanisms. FEBS Lett. 2016;590 (17):2888 - 2901.
[6] Barrera LA, Alvarez-Varela A, Kazane K, et al. CRISPR screens in vivo for tissue-specific cancer drivers. Science. 2016;353 (6307):1160 - 1163.
[7] Tzelepis K, de Stanchina E, D'Arcangelo M, et al. CRISPR-Cas9 screens in cancer cells to identify novel drug targets and resistance mechanisms. FEBS Lett. 2016;590 (17):2888 - 2901.










