一文快速了解CRISPR文库筛选

什么是CRISPR文库筛选?
CRISPR文库筛选 (CRISPR library screening)是一种基于 CRISPR/Cas9 基因编辑系统的高通量功能基因筛选技术,广泛应用于基因功能研究、疾病机制解析及药物靶点发现等领域。该技术通过合成覆盖目标基因组或特定功能基因集合的单导 RNA(single guide RNA,sgRNA)文库,并将其克隆至慢病毒等递送载体中,在低感染复数(MOI,通常 <0.3)的条件下转导至稳定表达 Cas9(或 dCas9 衍生系统)的细胞中,从而确保单个细胞内原则上仅整合一条 sgRNA,对应单一基因扰动事件。完成文库转导后,细胞群体通常被分为实验组和对照组。实验组在特定筛选压力条件下培养,例如小分子药物处理、营养或氧限制、免疫因子刺激、病毒感染等,而对照组维持基础培养条件。随着筛选过程的进行,对筛选压力敏感的细胞逐渐被淘汰,而具有耐受性或优势表型的细胞则被选择性富集。筛选结束后,通过提取两组细胞的基因组 DNA,对整合的 sgRNA 序列进行 PCR 扩增并结合高通量测序(NGS)分析,比较筛选前后或实验组与对照组中 sgRNA 的丰度变化,即可系统性地鉴定与特定表型或生物学过程相关的关键功能基因。与传统的 cDNA 过表达文库或 RNA 干扰(RNAi)筛选相比,CRISPR 文库筛选在基因扰动层面更接近真实的功能缺失或调控状态,具有敲除效率高、背景噪音低、可重复性好以及可扩展性强等优势,已成为当前功能基因组学研究中应用最为广泛和成熟的筛选平台之一。
CRISPR文库的类型
根据设计策略和应用目的的不同,CRISPR 文库可从多个维度进行分类。合理选择文库类型是保障筛选实验成功与结果可靠性的关键前提:
1.按覆盖范围分类:全基因组文库与亚文库
- 全基因组文库(Genome-wide library): 全基因组 CRISPR 文库针对特定物种的全部编码基因(protein-coding genes)设计 sgRNA,通常为每个基因配置多条 sgRNA,以提高基因扰动的覆盖度和统计可靠性。该类文库覆盖范围广,适用于未知机制探索、系统性靶点发现等研究场景,但整体规模较大,对细胞数量、病毒制备和数据分析能力要求较高。目前,针对人、小鼠、绿猴等常用研究模型的全基因组 CRISPR 文库已较为成熟,可直接用于体外或体内高通量筛选实验。
- 亚文库(Sub-library / Focused library): 亚文库通常围绕特定研究目标构建,针对某一基因家族、信号通路或功能类别进行定向设计,例如激酶家族、转录因子、代谢相关蛋白、细胞周期调控因子等。与全基因组文库相比,亚文库规模更小、筛选复杂度更低,更适合假设驱动型研究或验证性筛选。
在实际应用中,研究者可根据研究问题的明确程度,在全基因组文库与功能亚文库之间进行权衡。对于目标明确的研究方向,优先选择高覆盖度、设计成熟的现货亚文库,有助于降低实验成本并提升筛选效率。基于不同研究需求,源井生物可支持多物种、不同覆盖范围的 CRISPR 文库设计与构建方案。
2.按筛选目的分类:功能缺失型文库与功能获得型文库
-
功能缺失型文库(Loss-of-Function,LOF)
LOF 筛选旨在通过降低或消除基因功能来评估其在特定表型中的作用,主要包括以下两类:- CRISPR-KO 文库: 利用 Cas9 核酸酶在靶基因编码区引入双链断裂(DSB),经非同源末端连接(NHEJ)修复后产生插入或缺失突变,从而实现基因的不可逆敲除。CRISPR-KO 文库适用于识别非必需基因或筛选对细胞生存、耐药性等具有显著影响的功能基因。
- CRISPRi 文库(CRISPR interference): 采用催化失活的 dCas9 蛋白融合转录抑制结构域(如 KRAB),靶向基因启动子或转录起始位点附近区域,对基因转录进行可逆性抑制。相较于 KO 策略,CRISPRi 更适用于必需基因(完全敲除导致细胞死亡)以及非编码区或调控元件的功能研究,在表型控制精度和安全性方面具有优势。
- 功能获得型文库(Gain-of-Function,GOF): GOF 筛选主要通过增强基因表达来研究其功能,通常采用 CRISPRa 文库(CRISPR activation)。 该体系利用 dCas9 融合转录激活结构域(如 VP64、p65、Rta 等),靶向基因启动子区域,从而上调内源基因表达。CRISPRa 文库在研究基因过表达效应、筛选耐药或促生长因子等方面具有重要应用价值。
在实际项目中,LOF 与 GOF 文库常根据研究目标进行互补使用。源井生物在文库设计阶段可结合基因特性和实验模型,协助研究者选择更合适的功能扰动策略。
3.其他特殊类型文库
除传统 KO、CRISPRi 和 CRISPRa 文库外,随着基因编辑技术的发展,还衍生出多种面向特定研究需求的特殊文库类型:
- 条形码文库(Barcoded library): 在 sgRNA 表达载体中引入唯一分子条形码(barcode),可在结合单细胞转录组测序或谱系追踪分析时,实现对 sgRNA 与细胞表型的一一对应,适用于高分辨率异质性研究。
- 碱基编辑文库(Base editing library): 结合胞嘧啶碱基编辑器(CBE)或腺嘌呤碱基编辑器(ABE),针对特定位点构建单碱基突变文库,用于模拟疾病相关突变、研究功能性 SNP 或精细解析蛋白关键位点功能。这类文库无需产生双链断裂,在研究非显性表型和精细功能调控方面具有独特优势。
在实际研究中,往往需要结合研究目标、模型系统和筛选条件,对文库类型进行综合选择。源井生物可基于不同应用场景,支持从文库策略设计到实验实施的整体解决方案,助力研究者高效推进 CRISPR 文库筛选项目。
CRISPR文库筛选流程
CRISPR文库筛选的典型流程可分为以下几个步骤:
- 文库构建: 针对目标物种的每个基因设计多条sgRNA(常为3–6条/基因),使用高通量芯片合成对应寡核苷酸。通过Gibson酶法将这些寡核苷酸克隆至慢病毒载体(如lentiCRISPR)中,构建质粒文库。
- 病毒包装与转导: 将质粒文库混合后包装为慢病毒颗粒,并以低MOI(<0.3)感染Cas9表达的靶细胞系。这样保证大部分细胞仅获得一个gRNA载体。细胞数量通常为sgRNA总数的200–1000倍以上,以保证文库覆盖度和均匀性。
- 细胞筛选: 将感染后得到的细胞文库分成两组,一组作为对照组,一组作为实验组。在实验组中施加特定筛选压力(如药物处理、缺氧、病毒感染或其它生物学刺激)。在这一“优胜劣汰”过程中,对压力敏感的细胞会死亡或生长减慢,而更耐受的细胞会增殖富集。
- NGS测序与数据分析: 对实验组和对照组细胞分别提取基因组DNA,利用PCR扩增整合到基因组中的sgRNA序列段,然后进行高通量测序。通过比对测序reads至文库参考序列并统计sgRNA的丰富度变化,可以识别出与筛选条件相关的候选基因。
- 候选基因验证: 对筛选得到的潜在靶点进行二次验证,如使用单个gRNA敲除或构建基因过表达/回补细胞系,观察对应的表型变化,以确认其功能关联。
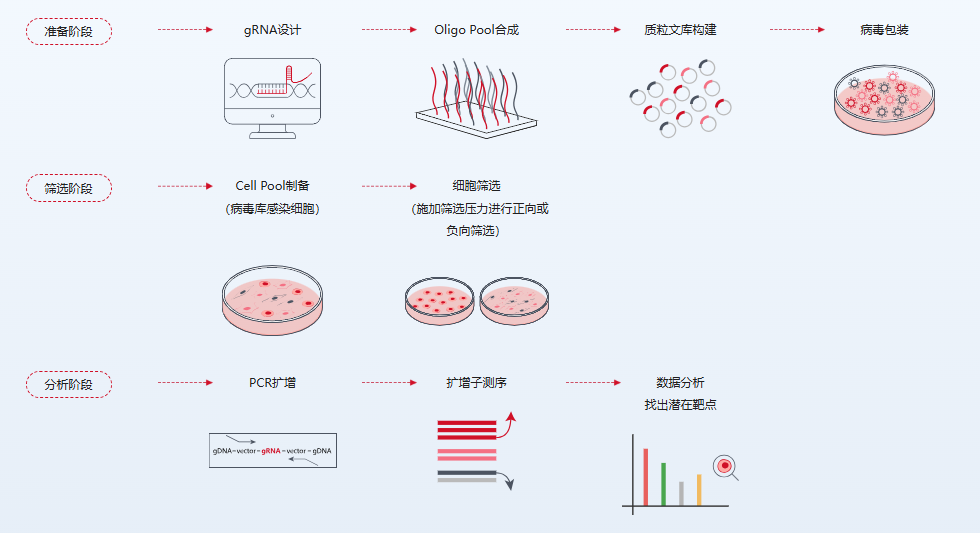
图1. CRISPR文库筛选流程示意图。示意图中展示了从gRNA设计、文库构建到病毒感染、细胞筛选与测序分析的各主要步骤。在筛选过程中,按照“优胜劣汰”的原则,耐受筛选压力的细胞被富集,而敏感的细胞则被淘汰,从而通过比较筛选前后各gRNA的丰度差异来定位关键基因。
上述流程主要适用于CRISPR文库体外(in vitro)筛选。在CRISPR文库体内(in vivo)筛选中,研究者通常需要将文库导入活体环境:可以先在体外构建好携带CRISPR文库的细胞系,然后将其移植到小鼠体内,或者直接利用AAV/腺病毒载体在动物体内递送CRISPR文库。体内筛选能够模拟机体完整的免疫系统、血管网络和组织微环境,使结果更贴近生物体实际状态。
CRISPR文库体外筛选流程
CRISPR 文库体外筛选 (in vitro CRISPR library screening)是在受控的细胞培养体系中开展的大规模功能基因筛选,是目前应用最为成熟、也是最常用的筛选形式之一。在标准的体外筛选流程中,通常按照 CRISPR 文库筛选的通用步骤开展实验:首先完成 sgRNA 文库的设计与构建,并将其包装为慢病毒载体;随后在低感染复数(MOI)条件下将文库转导至稳定表达 Cas9(或 dCas9 衍生系统)的细胞系中,建立具有充分覆盖度的细胞文库。文库细胞经抗性筛选或稳定培养后,即可进入功能筛选阶段。在筛选阶段,细胞通常在细胞培养皿或多孔板中进行培养,并根据研究目的施加特定筛选压力。常见的体外筛选条件包括小分子药物或候选化合物处理、培养环境改变(如缺氧、营养限制或代谢应激)、免疫或炎症相关刺激,以及病毒或细菌感染等。通过在体外条件下对细胞施加可控且可重复的选择压力,可以系统性评估基因扰动对细胞生存、增殖或功能表型的影响。随着筛选过程的推进,对筛选条件敏感的细胞逐渐被淘汰,而具有适应性或优势表型的细胞群体得到相对富集。筛选结束后,可根据具体实验设计,采用流式细胞术分选、细胞存活率或增殖能力评估、长期培养代数变化等方式对细胞群体进行进一步富集或分组处理。随后提取不同实验组细胞的基因组 DNA,对整合的 sgRNA 序列进行 PCR 扩增并结合高通量测序分析,从而识别在筛选过程中显著富集或耗竭的 sgRNA 及其对应的功能基因。
CRISPR 文库体外筛选具有实验条件可控性强、重复性高、筛选周期相对较短以及易于放大实验规模等优势,特别适用于早期功能基因筛选、作用通路解析和候选靶点的初步发现阶段。基于成熟的细胞模型和稳定的筛选体系,源井生物可支持不同研究场景下的体外 CRISPR 文库筛选实验设计与实施,为后续体内验证或机制研究提供可靠的功能基因基础。
CRISPR文库体内筛选流程
CRISPR 文库体内筛选 (in vivo CRISPR library screening)是在完整生物体环境中开展的功能基因筛选方式,能够在保留组织结构、微环境及系统性调控因素的前提下,系统解析基因在复杂生理或病理状态中的功能作用。在典型的体内筛选流程中,首先在体外构建并充分验证携带 CRISPR 文库的细胞文库cell pool,包括文库覆盖度、sgRNA 分布均一性及基因编辑效率等关键质量指标。随后,将文库细胞通过皮下接种、原位移植或血管注射等方式引入实验动物体内,常用模型包括免疫缺陷小鼠或特定遗传背景的小鼠模型,以避免免疫排斥对筛选结果的干扰。
移植完成后,细胞在动物体内经历自然生长、分化或迁移过程,形成肿瘤组织或特定靶器官定植模型。在此基础上,可根据研究目的施加相应的体内筛选压力,例如药物给药、免疫相关干预、代谢或炎症刺激等,以模拟真实疾病发生和进展过程。随着筛选周期的推进,具有适应性优势的基因扰动事件将在体内环境中逐步富集。筛选终点通常通过处死动物并采集肿瘤组织或目标器官来完成。随后从组织样本中分离细胞,提取基因组 DNA,对整合的 sgRNA 序列进行 PCR 扩增并结合高通量测序(NGS)分析,以比较不同实验条件下 sgRNA 的丰度变化,从而鉴定与体内表型密切相关的候选功能基因。体内 CRISPR 文库筛选能够更真实地反映基因功能与组织微环境、免疫调控、血管系统等多因素之间的相互作用。例如,在肿瘤转移研究中,可将携带 CRISPR 文库的肿瘤细胞经血管注射至小鼠体内,通过分析远端转移灶中的 sgRNA 富集情况,筛选出参与肿瘤侵袭和转移过程的关键调控基因。
相较于体外筛选,体内筛选在技术和实施层面也面临更高挑战,包括文库递送效率受限、筛选周期较长、实验成本较高以及动物伦理和实验设计复杂性等问题。因此,体内筛选通常需要在前期经过充分的体外验证,并结合严谨的实验设计和统计分析策略进行实施。基于对体内模型和筛选流程的系统理解,源井生物可支持从文库构建到体内筛选实施及后续数据分析的整体技术方案,为复杂表型研究和靶点验证提供可靠支撑。
CRISPR文库数据分析
CRISPR 文库筛选实验产生的数据规模大、结构复杂,其数据分析环节是整个筛选流程中至关重要的一步,直接影响候选基因识别的准确性和生物学结论的可靠性。由于筛选结果本质上依赖于不同条件下 sgRNA 丰度变化的统计比较,如何进行合理的数据质控、建模和结果解读,始终是 CRISPR 文库应用中的技术难点之一。目前,CRISPR 文库筛选领域已发展出多种成熟的生物信息学分析工具,包括 MAGeCK / MAGeCK-VISPR、ScreenBEAM、BAGEL、CasTLE、CRISPRcleanR、JACKS 等。这些工具在统计模型、噪声处理和基因评分策略上各有侧重,适用于不同类型的筛选实验(如正向筛选、负向筛选或时间序列分析)。研究者通常需要结合实验设计和数据特征,选择合适的分析软件,或通过多种算法交叉验证以提升结果的稳健性。
以应用较为广泛的 MAGeCKFlute 分析流程为例,其典型分析步骤包括:
- 原始测序数据质控(Quality Control): 对高通量测序数据进行质量评估,过滤低质量 reads,并准确提取 sgRNA 序列,确保后续分析基于可靠数据。
- sgRNA 比对与计数: 将处理后的 reads 比对至参考 sgRNA 文库序列,统计每条 sgRNA 在不同样本或处理条件下的 reads 数,形成 sgRNA 丰度矩阵。
- 差异丰度分析与基因打分: 基于负二项分布等统计模型,对实验组与对照组之间的 sgRNA 丰度变化进行比较,识别在筛选条件下显著富集(正向调控)或显著耗竭(负向调控)的 sgRNA,并进一步整合至基因层面,获得候选功能基因列表。
- 功能注释与通路分析: 对筛选得到的候选基因进行 GO、KEGG 等功能富集分析,并结合热图、火山图、排名图等可视化方式,辅助研究者从系统层面理解其潜在生物学意义。
需要注意的是,不同分析工具在统计假设、噪声校正和 sgRNA 整合策略上的差异,可能导致对同一数据集的敏感性和特异性存在一定偏差。因此,在实际研究中,结合多种分析方法进行交叉验证,已成为提升 CRISPR 文库筛选结果可信度的常见策略。
基于对 CRISPR 文库筛选数据特点和分析流程的系统理解,源井生物推出 iScreenAnlys™ CRISPR 文库分析平台, 旨在为研究者提供标准化、自动化的数据分析支持。该平台在分析流程层面深度集成主流统计方法(如 MAGeCK 分析框架)与数据质控模块,可自动完成从 sgRNA 计数、统计分析到基因层级整合与结果标注的完整流程,并输出符合国际期刊发表规范的可视化图表与分析结果。在此基础上,平台支持多维度参数配置和结果展示,适用于不同类型的 CRISPR 文库筛选实验设计。通过将复杂的分析流程进行模块化和流程化封装,iScreenAnlys™ 平台在一定程度上降低了 CRISPR 文库数据分析的技术门槛,使缺乏编程或生物信息学背景的研究人员也能够高效完成筛选数据解析,从而更加专注于生物学问题本身。该平台可作为 CRISPR 文库筛选实验的重要数据分析工具,辅助研究者系统挖掘候选基因并提升筛选研究的整体效率。
CRISPR文库应用案例
1. 癌症治疗
1)识别肿瘤必需基因,针对致癌基因进行靶向治疗。
恶性肿瘤累积了很多基因突变,但这些肿瘤细胞维持恶性表型通常只依赖其中某个/某些突变活化的癌基因,这种现象叫 癌基因成瘾 (oncogene addiction),这些癌基因也称为 驱动癌基因 (driver oncogenes)。阻断这些驱动基因的活性可以诱导肿瘤细胞发生快速调亡、生长阻滞或分化,而对正常细胞影响较小,这种差异化为肿瘤的 靶向治疗 带来了希望。
近年来二代测序的广泛应用已经找出很多肿瘤细胞存在的突变点,但是无法很好的区分哪些是驱动突变。通过基因失活找出肿瘤细胞生存依赖的驱动基因是个更简单直接的方法。 CRISPR文库可以同时靶向全基因组不同基因,是实现高通量基因失活的绝佳工具。 2014年,Shalem等[1]首次利用GeCKO文库鉴定出人黑色素瘤细胞和多能干细胞存活的关键基因(图1)。Wang等[2]利用CRISPR全基因组文库筛选将NCAPG鉴定为肝细胞癌肿瘤生长的必须癌基因。Kiessling等[3]利用CRISPR全基因组文库筛选验证了HCC-827细胞的驱动基因EGFR和CHP-212细胞的驱动基因NRAS和MEK1,同时发现了新的驱动基因TBK1和TRIB2并用于进一步研究。
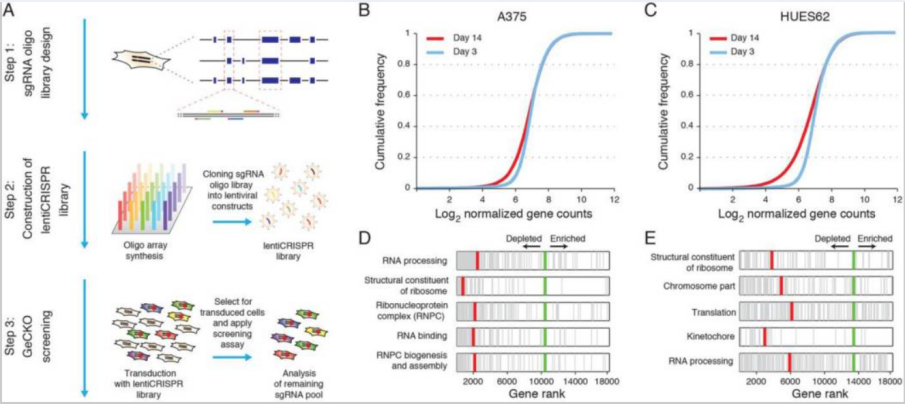
图2.利用CRISPR文库筛选A375与HUES62存活必需基因
2)筛选合成致死作用靶点,实现精准治疗。
针对致癌基因可以使用抑制剂进行靶向治疗,而对于抑癌基因而言,其功能的失活导致无法直接靶向,通常采用靶向与失活的抑癌基因具有合成致死作用的基因搭档。 合成致死(Synthetic lethality) 指两个非致死基因同时失活导致细胞死亡的现象。假如肿瘤细胞中存在特定基因A失活,那么用药物抑制它的合成致死搭档基因B,使两者都失活,而健康的体细胞因为有正常的基因A,能够保证正常的生理功能的表达,不会受到药物的伤害,从而只特异性的杀死该类肿瘤细胞(如图3)[4]。
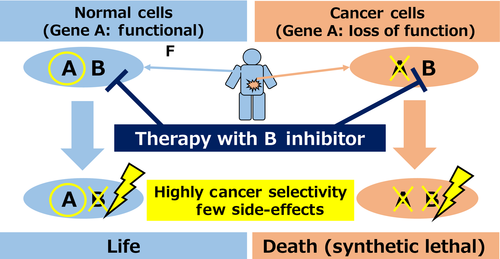
图3.合成致死疗法
合成致死最典型的应用就是PARP抑制剂治疗BRCA1/2突变的肿瘤,截止2020年,PARP抑制剂全球销售额已经超过20亿美元!那么如何寻找更多具有合成致死作用的基因对呢?CRISPR文库高通量筛选起了大作用!
CRISPR文库筛选合成致死作用一般有两种逻辑, 一种是直接设计双重打靶的gRNA载体,即在一个细胞内同时靶向两个基因 ,筛选具有合成致死作用的基因对, Shen等[5]采用这种方法对3种实验癌细胞系(Hela、A549和293T)中的73个基因进行了150000个组合的筛选,找出120种有合成致死作用的基因对(如图3)。 另外一种逻辑就是在单个抑癌基因敲除的细胞系上进行文库筛选 ,筛出与敲除的抑癌基因具有合成致死作用的靶点,Feng等[6]就是采用这种方法先在293A细胞上构建了12个已知抑癌基因敲除细胞系,再用全基因组敲除文库对野生型和敲除细胞系进行CRISPR筛选,分析同一细胞筛选前后gRNA的丰度变化,通过对比野生型和抑癌基因敲除细胞系在CRISPR筛选后gRNA的丰度差异确定潜在的合成致死基因。
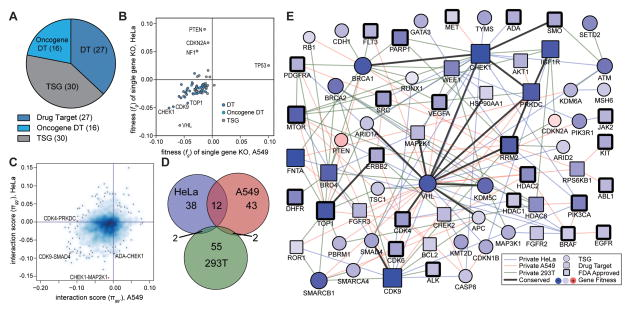
图4.利用CRISPR文库筛选基因相互作用关系
3)寻找肿瘤耐药相关基因,研究耐药机制,优化治疗方案。
肿瘤分子靶向药物面临的主要问题是耐药,因此探讨靶向药物的耐药机制有着重要的临床意义。经CRISPR文库正向筛选,绝大部分细胞因对药物敏感而死亡,部分细胞由于发生特定基因敲除产生抗药性而存活了下来。通过检测存活细胞携带的gRNA可以将与耐药性产生相关的基因筛选出来。
Vemurafenib被批准用于携带BRAFV600E 突变的晚期黑色素瘤的治疗。为了研究Vemurafenib的耐药机制,Shalem等[1]设计了包含64751条sgRNA、靶向全基因组18080个基因的敲除文库,经过细胞感染,加药富集,最终筛选出nf1、med12、nf2、cul3、tada2b和tada1等耐药相关基因,并提出了新的Vemurafenib肿瘤耐药机制假说。
肿瘤耐药领域更多文库筛选应用,可点击↓
>>>CRISPR文库锁定自噬相关基因,找出肝癌耐药“元凶”
2.筛选病毒感染相关的宿主因子
CRISPR文库筛选还常用于研究病毒感染相关的宿主因子。针对丙肝病毒、登革病毒、日本脑炎病毒和西尼罗病毒,目前已经筛选出了一批与内质网功能以及受体相关的宿主因子。
如,Zhang等人[7]通过CRISPR/Cas9 的全基因组筛选验证了黄病毒感染所需的九个人类基因,这些基因都与内质网 (ER) 功能(包括易位、蛋白质降解和 N-连接糖基化)相关。Ma等人[8]通过设计包含77,406个sgRNAs、针对20,121个基因的文库,对全基因组进行筛选,得出EMC2、EMC3、SEL1L等7个附属于内质网相关蛋白降解通路(ERAD)的基因。进一步实验表明,敲除这些基因后不会阻止西尼罗病毒(WNV)的复制,但会阻止该病毒致死细胞。
病毒领域更多文库筛选应用,可点击↓
>>>CRISPR文库:为病毒研究打开新视角——初阶干货(一)
3.信号通路、抗体靶标等
除了肿瘤相关基因筛选和病毒感染宿主因子筛选,CRISPR文库筛选还在抗体靶标筛选、细胞信号通路研究、非编码基因功能研究等方面有着广泛的应用。
挑战与优化建议
尽管 CRISPR 文库筛选在功能基因组学研究中展现出巨大潜力,但在实际应用过程中仍面临多方面的技术挑战。对这些问题的充分认知与系统优化,是保障筛选结果可靠性和可重复性的关键。
- 脱靶效应与背景噪声: 尽管现代 CRISPR 文库在 sgRNA 设计阶段已充分考虑序列特异性,但非零脱靶切割仍不可完全避免,可能在一定程度上干扰筛选结果的准确性。此外,sgRNA 活性差异、编辑效率不均以及文库构建和扩增过程中的偏倚,均可能引入背景噪声。另一方面,细胞群体本身的异质性也会对功能获得型或功能缺失型筛选结果产生影响,使部分基因扰动在不同细胞亚群中呈现出差异化表型,从而增加结果解读的复杂度。
- 载体递送效率与模型限制: 慢病毒载体在多数常规细胞系中具有较高的递送效率,但在原代细胞、干细胞或某些特殊组织来源细胞中,其转导效率和稳定性往往受到限制。同时,在体内筛选场景下,病毒递送还可能受到组织屏障、免疫反应及分布不均等因素影响。尤其是在体内 CRISPR 文库筛选中,难以保证文库中所有 sgRNA 在目标组织或细胞中的均匀递送和表达,容易导致文库覆盖度不足,从而影响筛选结果的代表性和统计学可靠性。
- 多基因网络与协同效应的复杂性: 许多生物学过程并非由单一基因调控,而是依赖多基因之间的协同作用和调控网络。针对双基因或多基因相互作用开展筛选,通常需要构建规模庞大的组合型文库,这在实验实施、样本规模和数据分析层面均提出了更高要求。目前多数分析工具仍以单基因效应为核心假设,对多因子协同效应的建模能力有限,难以系统性覆盖所有潜在基因组合,从而在一定程度上限制了复杂遗传网络的全面解析。
针对上述挑战,可从以下方向进行优化:采用带条形码或可诱导元件的文库设计,并结合单细胞转录组测序,实现细胞谱系追踪和异质性解析。开发新型递送系统,如整合空间转录组学技术的纳米载体,以提高体内筛选的递送效率和靶向性。进一步优化sgRNA设计策略(如优先靶向保守结构域)并利用深度学习等人工智能方法构建基因相互作用网络,从而简化文库规模并挖掘协同调控机制。同时,建立标准化的数据库和分析流程,促进多组学数据整合,为复杂表型的机制解析提供支持。随着这些技术的不断改进,CRISPR文库筛选的精度和效率将显著提升。
总结与展望
CRISPR 文库筛选凭借其高通量、可编程和高度可扩展的技术优势,已成为功能基因组学研究和药物靶点发现领域中的核心工具之一。通过在基因组尺度上系统性扰动基因功能,该技术为复杂生物过程的机制解析提供了前所未有的研究手段,并在肿瘤生物学、病毒感染机制、免疫调控及细胞信号通路研究等多个方向取得了重要进展。
随着技术体系的不断演进,CRISPR 文库筛选正逐步与单细胞测序、空间组学及人工智能等前沿技术深度融合。单细胞层面的解析能力使研究者能够更精细地刻画细胞异质性和动态变化,而空间组学技术则为理解基因功能在组织微环境中的空间分布提供了新的视角。同时,基于机器学习和深度学习的数据分析方法,正在推动筛选结果从“基因列表”向“调控网络”和“功能模块”层面转化。
在实验层面,更高效、更精准的递送系统以及不断优化的文库设计策略,也将进一步拓展 CRISPR 文库筛选的应用边界。近年来的研究表明,直接在动物体内开展的 CRISPR 文库筛选能够揭示许多体外模型中难以观察到的基因依赖关系和调控机制,为理解真实生理或病理状态下的基因功能提供了重要补充。
展望未来,体内与体外筛选策略的有机结合、多样化文库体系的灵活应用,以及智能化数据分析工具的持续发展,将显著提升 CRISPR 文库筛选在复杂表型研究和转化医学中的价值。总体而言,CRISPR 文库筛选技术在精准医学和功能基因组学领域仍具有广阔的发展空间,有望在更深层次上推动对生命过程和疾病机制的系统性理解。
关于源井生物
源井生物围绕CRISPR功能基因组学研究,构建了系统化的文库筛选技术体系。其自主研发的 CRISPR-iScreen™ 技术平台, 旨在提升 CRISPR 文库筛选在设计、实施及数据分析层面的整体效率,为功能基因筛选和靶点发现提供稳定可靠的技术支持。目前,源井生物已建立覆盖多个物种和应用场景的 CRISPR 文库资源体系,现货文库种类超过 40 种,涵盖人类、小鼠、绿猴及猪等常用研究模型的 全基因组基因敲除(CRISPR-KO)、抑制(CRISPRi)和激活(CRISPRa)文库。 在此基础上,源井生物还构建了多种功能导向型 CRISPR 亚文库,针对激酶、细胞周期调控因子、膜蛋白、代谢相关基因等特定基因集合,支持更聚焦、更高效的功能筛选研究。源井生物可提供多样化的文库现货资源,包括 150余种文库病毒现货及400余种CRISPR文库Cell Pool现货, 以满足不同实验模型和研究阶段对CRISPR文库的快速应用需求。
同时,源井生物提供覆盖体外与体内CRISPR文库筛选的一站式功能筛选服务,从文库策略设计、实验实施到数据分析形成完整技术闭环。配套的 iScreenAnlys™ 文库分析平台,集成 Drug-Z、MAGeCK-MLE 及 MAGeCK-RRA 等主流统计分析算法,可实现 CRISPR 筛选数据的标准化处理与一键分析,辅助研究者高效挖掘候选功能基因,加速研究进程。
| 产品名称(中文) | 靶向基因 | gRNA数量 | 非靶标对照sgRNA数量 | 病毒包装系统 | 质粒系统 |
|---|---|---|---|---|---|
| 人全基因组敲除文库(单质粒系统A) | 19050 | 65383 | 1000 | 第三代病毒包装系统 | 单质粒系统 |
| 人全基因组敲除文库(单质粒系统B) | 19050 | 58028 | 1000 | 第三代病毒包装系统 | 单质粒系统 |
| 人全基因组敲除文库(单质粒系统A+B) | 19050 | 123411 | 1000 | 第三代病毒包装系统 | 单质粒系统 |
| 人全基因组敲除文库(双质粒系统A) | 19050 | 65383 | 1000 | 第三代病毒包装系统 | 双质粒系统 |
| 人全基因组敲除文库(双质粒系统B) | 19050 | 58028 | 1000 | 第三代病毒包装系统 | 双质粒系统 |
| 人全基因组敲除文库(双质粒系统A+B) | 19050 | 123411 | 1000 | 第三代病毒包装系统 | 双质粒系统 |
| 小鼠全基因组敲除文库(单质粒系统A) | 20611 | 67405 | 1000 | 第三代病毒包装系统 | 单质粒系统 |
| 小鼠全基因组敲除文库(单质粒系统B) | 20611 | 62804 | 1000 | 第三代病毒包装系统 | 单质粒系统 |
| 小鼠全基因组敲除文库(单质粒系统A+B) | 20611 | 130209 | 1000 | 第三代病毒包装系统 | 单质粒系统 |
| 小鼠全基因组敲除文库(双质粒系统A) | 20611 | 67405 | 1000 | 第三代病毒包装系统 | 双质粒系统 |
| 小鼠全基因组敲除文库(双质粒系统B) | 20611 | 62804 | 1000 | 第三代病毒包装系统 | 双质粒系统 |
| 小鼠全基因组敲除文库(双质粒系统A+B) | 20611 | 130209 | 1000 | 第三代病毒包装系统 | 双质粒系统 |
参考文献:
[1] Shalem O, Sanjana NE, Hartenian E, Shi X, Scott DA, Mikkelson T, Heckl D, Ebert BL, Root DE, Doench JG, Zhang F. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 2014 Jan 3;343(6166):84-87. doi: 10.1126/science.
[2] Wang Y, Gao B, Tan PY, Handoko YA, Sekar K, Deivasigamani A, Seshachalam VP, OuYang HY, Shi M, Xie C, Goh BKP, Ooi LL, Man Hui K. Genome-wide CRISPR knockout screens identify NCAPG as an essential oncogene for hepatocellular carcinoma tumor growth. FASEB J. 2019 Aug;33(8):8759-8770. doi: 10.1096/fj.201802213RR
[3] Kiessling M K, Schuierer S, Stertz S, et al. Identification of oncogenic driver mutations by genome-wide CRISPR-Cas9 dropout screening[J]. BMC genomics, 2016, 17(1): 1-16.
[4] Sasaki M, Ogiwara H. Synthetic lethal therapy based on targeting the vulnerability of SWI/SNF chromatin remodeling complex‐deficient cancers[J]. Cancer Science, 2020, 111(3): 774-782.
[5] Shen JP, Zhao D, Sasik R, Luebeck J, Birmingham A, Bojorquez-Gomez A, Licon K, Klepper K, Pekin D, Beckett AN, Sanchez KS, Thomas A, Kuo CC, Du D, Roguev A, Lewis NE, Chang AN, Kreisberg JF, Krogan N, Qi L, Ideker T, Mali P. Combinatorial CRISPR-Cas9 screens for de novo mapping of genetic interactions. Nat Methods. 2017 Jun;14(6):573-576. doi: 10.1038/nmeth.4225.
[6] Feng X, Tang M, Dede M, et al. Genome-wide CRISPR screens using isogenic cells reveal vulnerabilities conferred by loss of tumor suppressors[J]. Science advances, 2022, 8(19): eabm6638.
[7] Zhang R, Miner JJ, Gorman MJ, Rausch K, Ramage H, White JP, Zuiani A, Zhang P, Fernandez E, Zhang Q, Dowd KA, Pierson TC, Cherry S, Diamond MS. A CRISPR screen defines a signal peptide processing pathway required by flaviviruses. Nature. 2016 Jul 7;535(7610):164-8. doi: 10.1038/nature18625 .
[8] Ma H, Dang Y, Wu Y, Jia G, Anaya E, Zhang J, Abraham S, Choi JG, Shi G, Qi L, Manjunath N, Wu H. A CRISPR-Based Screen Identifies Genes Essential for West-Nile-Virus-Induced Cell Death. Cell Rep. 2015 Jul 28;12(4):673-83. doi: 10.1016/j.celrep.2015.06.049 .










