CRISPR 文库筛选数据分析深读:DrugZ 与 MAGeCK-RRA 的机制对比与选择

引言
在 CRISPR-Cas9 全基因组筛选(CRISPR Screening) 实验中,如何从数以万计的 sgRNA 测序数据中精准地识别出具有生物学意义的“候选基因(Hits)”,是数据分析中最关键的一环。基因层面的统计方法决定了结果的灵敏度与可靠性,其中 MAGeCK-RRA 和 DrugZ 是最广泛使用的两类算法。虽然两者实现目的相同·,但其底层的数学逻辑和对数据的“价值观”却截然不同。本文将从核心思路、基因评价策略以及结果解读三个维度,深入剖析这两种算法的差异,帮助研究人员根据实验目的选择最合适的工具。
一、 核心算法哲学:关注“等级” vs. 关注“强度”
这是两种算法最根本的分歧点,决定了它们对原始数据的敏感度不同。
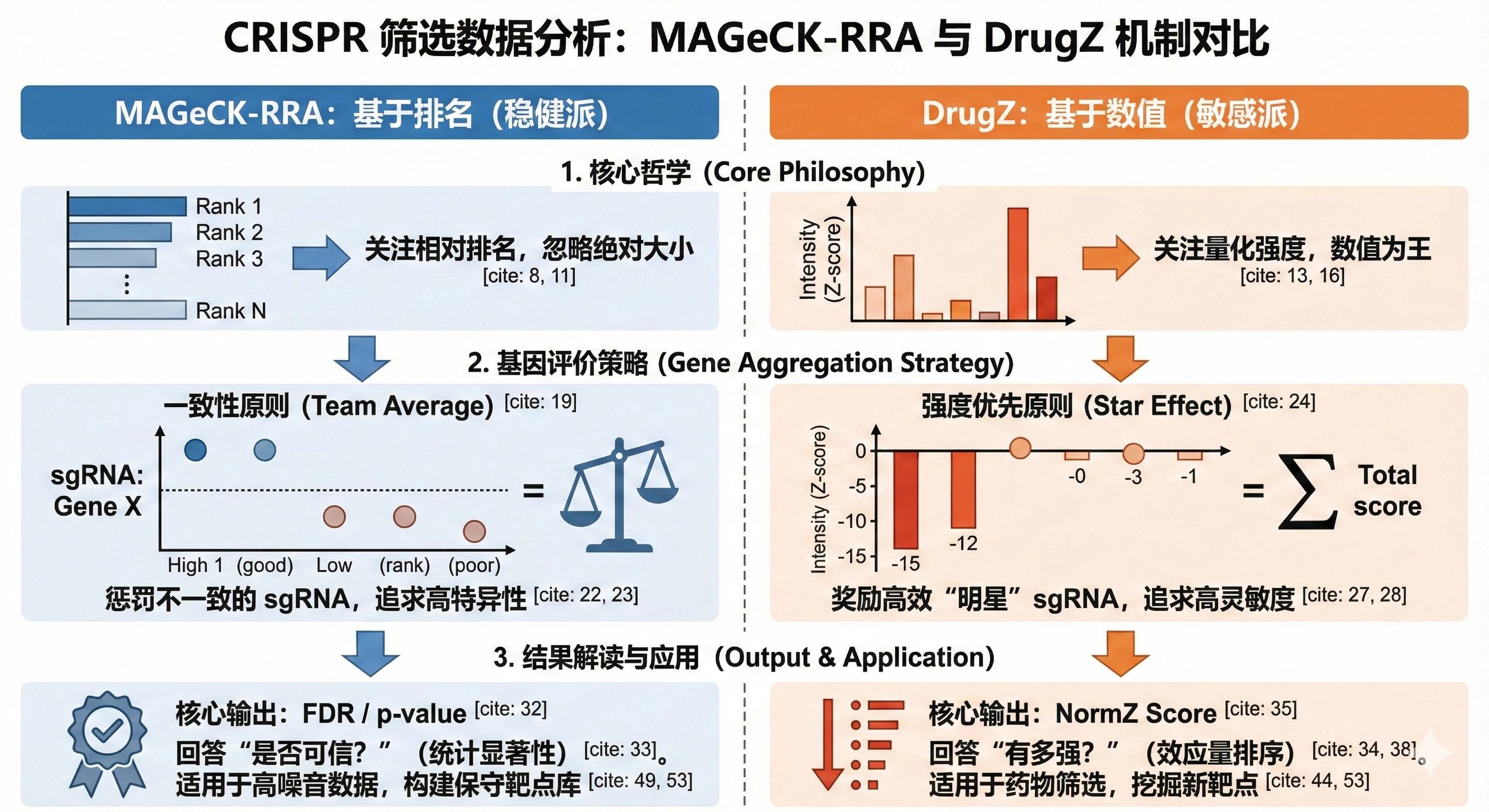
1. MAGeCK-RRA:基于排名的稳健派 (Non-parametric)
RRA (Robust Rank Aggregation) 的核心思想是基于 sgRNA 变化的相对顺序进行统计推断,而非依赖具体的变化倍数。可以简单的理解为RRA的逻辑是民主投票,排名优先。
- 工作机制: 它首先计算每个 sgRNA 在对照组和处理组之间的变化显著性,然后将所有 sgRNA 从变化最显著到最不显著进行排序(Rank)。
- 判定标准: 在评价一个基因是否重要时,RRA 会问:“靶向这个基因的大部分 sgRNA,是否都挤在排行榜的前列?”
- 主要特点: RRA 是一种非参数方法,核心特点在于关注排名的相对顺序,而非变化的绝对幅度。这使其对数据中的异常值和极端分布不敏感,增强了分析的稳健性。举例:假设 sgRNA-A 的丰度下降了 100 倍,sgRNA-B 下降了 10,000 倍。在 RRA 的排名系统中,它们可能紧挨着分别是第 1 名和第 2 名。RRA 认为它们“一样好”,从而忽略了 sgRNA-B 在生物学上可能代表着更强的致死性或表型。这种对**变化具体大小(Magnitude)**的忽略,导致了部分信息的丢失。
2. DrugZ:基于数值的强度派 (Parametric)
DrugZ 直接建立在正态分布假设下,以变化量本身作为主要信息来源。可以简单理解为它的核心逻辑是量化强度,数值为王。
- 工作机制: 它不进行排名,而是直接利用正态分布假设,计算每个 sgRNA 变化的 Fold Change,并将其转换为标准化的 Z-score(强度分) 。
- 判定标准: DrugZ 直接累加数值。它认为变化倍数本身蕴含了重要的生物学信息。
- 主要特点: 核心特点在于直接利用变化的幅度信息。这使得它能对效应强度进行量化,对由少数强效sgRNA驱动的基因信号更为敏感。一个变化了 10,000 倍的 sgRNA 会获得比变化 100 倍的 sgRNA 高得多的 Z-score(例如 -10 分 vs -2 分)。这使得 DrugZ 能够敏锐地捕捉到那些能够引发剧烈表型变化的基因,从而具有更高的灵敏度
二、 基因层面的综合评价:团队平均 vs. 明星效应
在真实的 CRISPR 筛选实验中,由于 sgRNA 设计效率、脱靶效应或染色质结构的差异,靶向同一个基因的 3-10 个 sgRNA 的效果往往是不均一的。两种算法处理这种“内部矛盾”的方式截然不同。
1. MAGeCK-RRA :一致性原则:追求高特异性
RRA 倾向于寻找那些“每个 sgRNA 表现都很一致”的基因。
- 场景模拟: 假设基因 X 有 5 个 sgRNA。其中 2 个效果极佳(排名极高),但另外 3 个由于设计效率低,效果平平(排名靠后)。
- 结果: 在 RRA 看来,这 3 个排名靠后的 sgRNA 是“拖后腿”的。因为 RRA 要求的是 整体排名的显著性, 这 3 个低排名会显著拉低基因 X 的评分(p-value 变大)。
- 总结: 这种基于一致性的策略虽然可能牺牲部分灵敏度,但能极为有效地 控制假阳性 ,确保结果的高度可靠。
2. DrugZ :强度优先原则:追求高灵敏度
DrugZ 更像是一个寻找“超级球星”的球探。它允许甚至鼓励个别 sgRNA 的突出表现。
- 场景模拟: 同样的基因 X,2 个强效 sgRNA 带来了巨大的负向 Z-score(例如 -15 分和 -12 分),而 3 个无效 sgRNA 的分数接近 0。
- 结果: DrugZ 计算的是总和(SumZ)。 $$(-15) + (-12) + 0 + 0 + 0 = -27$$ 即使有 3 个无效数据的稀释,那两个强效 sgRNA 贡献的巨大数值依然能支撑起整个基因的高分。
- 总结: DrugZ 极大地提高了 灵敏度。 它承认 sgRNA 效果不均一是实验常态,因此更容易发现那些由少数高效 sgRNA 驱动的真实阳性基因。这在药物筛选(Drug Screen)中尤为重要,因为药物处理下往往只有亲和力最高的 sgRNA 才会显示出剧烈变化。
三、 结果解读:可信度 vs. 效应量
当你拿到分析报告时,两种软件输出的核心指标也决定了你如何解读数据。
1. MAGeCK-RRA:“是否可信?”
- 核心指标: p-value 和 FDR。
- 解读: 它主要告诉你这个基因被筛选出来的结果 统计学上是否显著 ,即“这个结果是不是随机发生的?”。
2. DrugZ:“有多强?”
- 核心指标: normZ score (归一化 Z 分数)。
-
解读:
这个分数是 DrugZ 的精髓所在。
- 方向性: 负分(Negative)代表基因敲除后细胞对药物更 敏感 合成致死);正分(Positive)代表基因敲除后细胞更 耐药。
- 强度(Effect Size) 分数的绝对值直接对应表型的强弱。NormZ = -50 的基因比 NormZ = -10 的基因具有更强的生物学效应。
- 应用价值: 这让研究人员可以非常方便地进行 优先级排序(Prioritization) 。在后续验证实验资源有限的情况下,直接按照 NormZ 的绝对值从大到小挑选基因进行验证,往往能获得更高的阳性验证率。
四、 总结与建议
| 特性 | MAGeCK-RRA | DrugZ |
| 核心逻辑 | 非参数排名 (Ranking) | 参数化数值 (Z-score) |
| 对待极值 | 忽略具体大小,抗噪能力强 | 保留具体大小,容易受极值驱动 |
| 对 sgRNA 不一致的容忍度 | 低 (倾向于惩罚不一致) | 高 (倾向于奖励高效 sgRNA) |
| 灵敏度 | 较低 (保守,特异性高) | 较高 (善于发现新靶点) |
| 核心输出 | FDR / p-value | NormZ score |
| 需要注意的点 | 忽略了变化的强度。可能遗漏那些仅有部分强效sgRNA但生物学效应显著的基因 | 可能引入由单个异常高效sgRNA导致的假阳性信号(例如实验噪声过大时) |
什么时候选谁?
- 你的实验目的是药物筛选(Drug Screen)或合成致死筛选。
- 你需要尽可能多地找到潜在靶点(不想错过任何线索)。
- 你更关心基因功能的 效应强度 ,希望直接按重要性排序进行湿实验验证。
- 你确信你的数据噪音小,或者做了足够多组的生物学重复。
1.选择 DrugZ 的情况:
- 你的数据噪音极大,或者对照组样本测序深度不足。
- 你需要一个极其保守的列表,宁可漏掉,也不希望有假阳性。
- 作为 DrugZ 的补充验证,寻找两个算法的交集(Overlap),这通常是最稳健的候选列表。
2.选择 MAGeCK-RRA 的情况:
结论:
MAGeCK-RRA与DrugZ代表了两种不同但互补的数据分析哲学。RRA通过 一致性原则 提供了 高特异性 的结果,是构建可靠“核心靶点库”的基石;而DrugZ通过 强度优先原则 展现了 高灵敏度, 能有效扩展发现的边界,挖掘潜在的“隐藏靶点”。iScreenAnlys™ 文库分析平台是源井生物开发的交互式分析工具,支持多种主流统计方法(包括 MAGeCK-RRA、DrugZ、MLE 等)以及多类型的分析可视化输出,并提供灵活的个性化分析配置。平台旨在帮助科研人员以较低门槛完成 CRISPR 文库筛选数据的系统化处理与结果展示,适用于多种筛选实验场景。
源井生物基于自主研发的 CRISPR-iScreen™技术 ,可提供CRISPR-KO、CRISPRa、CRISPRi三大定制文库从高通量sgRNA文库构建到病毒包装、细胞转染、药物筛选、高通量测序和数据分析等一站式服务,多种交付方式满足不同科研需求。并可提供体内与体外 CRISPR 筛选服务,支持多种实验压力(如化合物处理、连续传代、病毒感染、流式分选等)和不同富集模式,适用于研究者自行设计的多类型功能筛选体系。CRISPR文库体外筛选低至1.5w。
如需了解或试用 iScreenAnlys™ 文库分析平台, 可与我们联系。>>










