CRISPR 文库筛选中的数据分析偏差:不同算法为何会得出截然不同的结果?

摘要
CRISPR文库筛选(CRISPR Screen) 是功能基因组学中非常重要的高通量工具,像一把多功能瑞士军刀,可以帮助科研人员找到致病基因、药物靶点以及耐药机制。然而,你可能没注意到:即便实验做得再完美,如果数据分析算法选错了,筛选结果可能会“跑偏”,让你白忙一场。不同的统计模型、归一化方法和多重检验策略,可能导致候选基因列表天差地别,甚至会直接影响科学结论的可靠性。今天,小源将介绍经典分析工具(MAGeCK、DESeq2/edgeR、PinAPL-Py)和新一代集成化平台 iScreenAnlys™文库分析平台,并结合实际实验条件提供策略性选型建议,帮助科研人员选择最合适的“分析小帮手”,降低实验风险,节省验证成本。
一、为什么数据分析在CRISPR文库筛选中如此关键?
想象一下,你在做一道数学题,计算器出错了,答案肯定不对。CRISPR文库筛选也是一样:实验设计再好,如果数据分析选错工具,结果可能完全偏离真实信号。
-
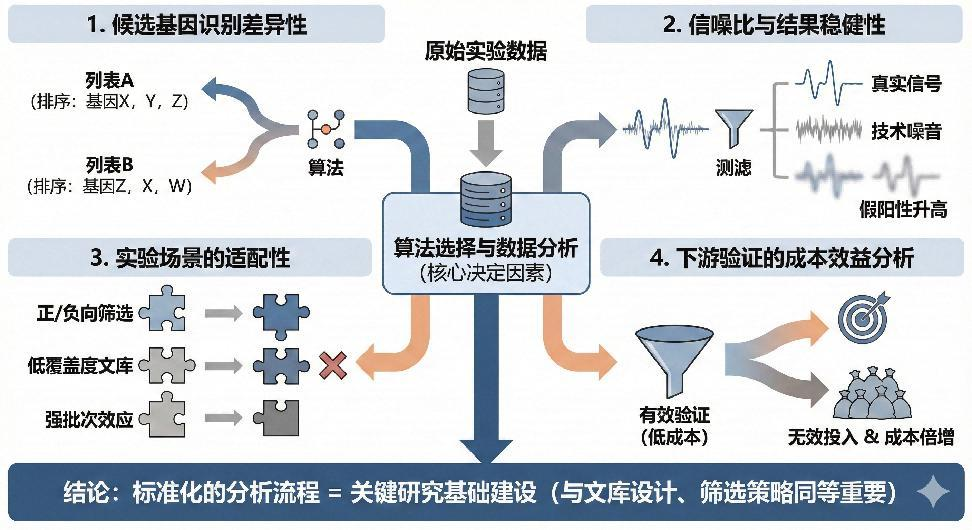
候选基因识别差异显著
不同算法就像不同品牌的计算器,同一份数据算出来的候选基因可能完全不同,甚至排序差异很大。这意味着,你本来以为很重要的基因,可能在另一种算法下被忽略。 -
信噪比与结果稳健性
一些算法像敏感雷达,能精准抓住真正的生物学信号;而有些算法容易被“技术噪音”干扰,假阳性增多,真阳性漏掉。选择不合适的工具,就像用劣质耳机听交响乐,细节全丢。 -
实验设计适配性
正向/负向筛选、文库覆盖度低、小重复数或者批次差异大,不同实验对算法的统计假设要求不同。算法不匹配,结果可能系统偏差,最终“误导”下游验证。 -
下游验证成本
筛选出来的几十到上百个候选基因,需要耗费大量时间和资金去做功能验证。分析阶段不严谨,可能会浪费大量实验资源,让人“心累”。
结论: 建立规范化、可复现的数据分析流程,与文库设计和实验策略同样重要,是确保科研效率和结果可靠性的关键。
二、 主流 CRISPR Screen 分析工具概览
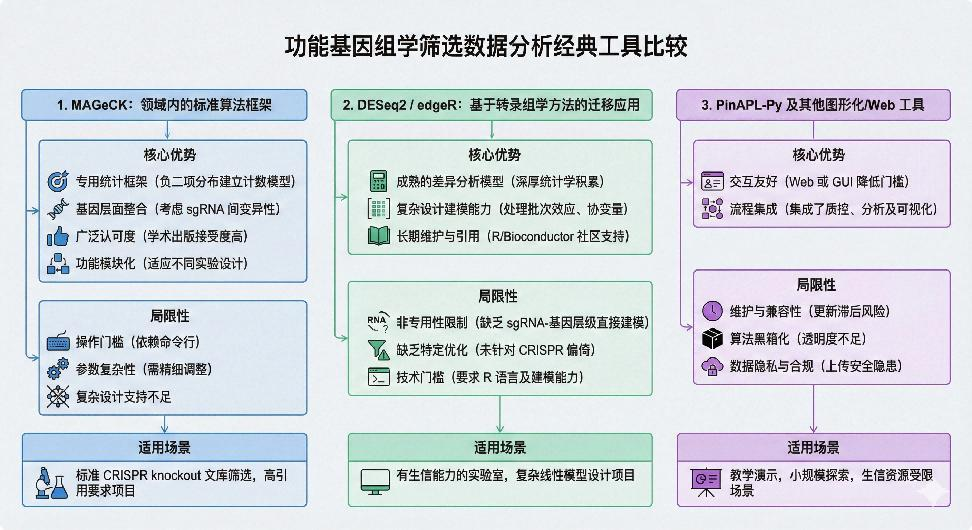
1. MAGeCK:CRISPR文库筛选的“老司机”
核心优势:
- 专用统计框架: 基于负二项分布建立计数模型,专为 CRISPR Screen 数据分布特征设计。
- 基因层面整合: 考虑 sgRNA 间的变异性,支持从 sgRNA 层面到基因层面的评分整合。
- 广泛认可度: 作为社区成熟工具,其分析结果在学术出版中具有极高的接受度。
- 功能模块化: 提供 mageck test 和 mageck mle 等模块,适应不同实验设计。
局限性:
- 操作门槛: 主要依赖命令行交互,对缺乏生物信息学背景的研究人员存在技术壁垒。
- 参数复杂性: 归一化、对照组设定及 sgRNA 过滤阈值等参数需要精细调整。
- 复杂设计支持不足: 在处理多因子复杂实验设计时,灵活性相对有限。
适用场景: 标准 CRISPR knockout 文库筛选;对结果引用权威性有较高要求的项目。
2. DESeq2 / edgeR:“转行高手”
核心优势:
- 成熟的差异分析模型: 在离散计数数据的负二项建模、归一化及方差估计方面具有深厚的统计学积累。
- 复杂设计建模能力: 通过设计矩阵(Design Matrix)有效处理批次效应、协变量及多因子交互设计。
- 长期维护与引用: R/Bioconductor 社区支持强大,文献引用广泛。
局限性:
- 非专用性限制: 本质为 RNA-seq 设计,缺乏对 sgRNA-基因层级关系的直接建模。
- 缺乏特定优化: 未针对 CRISPR 文库特有的偏倚、sgRNA 截尾(Truncation)等特性进行优化。
- 技术门槛: 参数、功能复杂,理解并使用难度较高,要求用户具备较强的 R 语言编程及统计建模能力。
适用场景: 拥有一定生信能力的实验室;涉及复杂线性模型设计的筛选项目;需与 RNA-seq 流程统一分析框架的研究。
3. PinAPL-Py 及其他图形化/Web 工具:“小白友好型”
核心优势:
- 交互友好: 提供 Web 或图形化界面(GUI),降低了操作门槛。
- 流程集成: 通常集成了质控、差异分析及可视化功能。
局限性:
- 维护与兼容性: 部分工具更新滞后,存在环境兼容性风险。
- 算法黑箱化: 参数与模型细节透明度不足,难以满足深度定制化分析需求。
- 数据隐私与合规: 公共 Web 平台可能存在数据上传的安全隐患。
适用场景: 教学演示;小规模探索性实验;生信资源受限且对数据隐私不敏感的场景。
三、源井iScreenAnlys™ 文库分析平台:一站式“分析神器”
在 CRISPR 文库筛选实验中,实验设计和文库构建固然重要,但数据分析往往是科研人员最头疼的环节——工具多、流程碎片化、操作复杂,团队协作也容易出问题。为解决这些痛点,源井生物自主研发了 iScreenAnlys™ 文库分析平台,提供真正意义上的一站式 CRISPR Screen 数据分析和可视化解决方案。平台特点可概括为:高度集成、即开即用、科研友好,帮助科研人员专注科学研究,而非工具操作。
iScreenAnlys™ 文库分析平台核心技术亮点:
-
1.端到端全流程集成(End-to-End Workflow)
iScreenAnlys™ 支持从原始计数矩阵或 FASTQ 测序数据导入开始,到数据质量控制(QC)、数据归一化、差异分析、可视化图标生成、分析报告导出的完整闭环流程。科研人员无需在多种工具之间来回切换,一键即可完成从数据到结果的全流程分析。 -
2. 针对 CRISPR 场景深度优化
平台内置适配 CRISPR 筛选特性的统计模型(如负二项回归、贝叶斯推断框架 等),并开放 R 包调用接口。可灵活支持正向/负向筛选、多条件比较等复杂实验设计。既满足初学者的易用需求,也兼顾高级用户的定制分析能力。 -
3. 强大的可视化与交互能力
一键生成并下载丰富图表,包括sgRNA 分布图、文库覆盖度分析、样本聚类、火山图及富集分析图表。图表美观直观,可直接用于论文、报告或演示,降低科研绘图压力。 -
4. 分析过程可追溯、可复现
系统可长期保存每次分析的全部结果及参数设置,后端审计机制确保分析流程 可复现、可追踪,满足论文审稿与项目存档的规范要求。科研团队再也不用担心“实验结果无法验证”的问题。 -
5. 协作友好、可扩展性强
支持多项目并行管理、数据在线共享,解决传统多人协作时文件混乱、版本不可控的问题。同时平台架构预留了扩展空间,可根据科研团队的需求接入更多模块,提升团队整体效率
设计理念: iScreenAnlys™ 文库分析平台并非意图取代经典算法,而是通过工程化封装,将已有成熟方法论进一步结构化、集成化与应用场景化。源井生物以“让基因编辑更简单”为目标,提升CRISPR文库筛选数据分析的效率、稳定性与可用性。简而言之,它是CRISPR文库筛选数据分析的 “智能助手”,让科研更高效、更直观、更可控”。
四、 CRISPR文库筛选分析工具选型的关键指标
在选择 CRISPR 文库筛选分析工具时,仅看功能是不够的。科学家们通常会通过量化指标评估工具的实际表现,确保筛选结果可靠、可复现。以下四类指标尤为重要:
1. 灵敏度与特异性(Sensitivity & Specificity)
- 灵敏度: 评估算法识别真实阳性基因(True Positives)的能力。
- 特异性: 评估算法排除技术噪音与假阳性(False Positives)的能力。
- 评估方法: 利用已知“金标准基因集”(如确定的药物靶点、核心信号通路)进行基准测试,计算候选基因列表的重叠率。
- 核心思路: 高灵敏度保证不漏掉关键基因,高特异性保证筛选结果靠谱。
2. 假发现率控制(FDR Control)
- FDR(False Discovery Rate): 高通量筛选统计中最重要的指标之一,用于控制假阳性的比例。
- 控制策略: 现代分析工具应集成 Benjamini-Hochberg 等标准校正算法,同时允许用户根据探索性或验证性目的灵活设定 FDR 阈值。
- 可视化辅助: 利用火山图等可视化手段,标记临界值附近的基因,辅助研究者结合生物学背景进行综合判读。
- 核心思路: 既要发现尽可能多的真实信号,又要避免太多假阳性浪费验证资源。
3. 归一化策略的稳健性(Robust Normalization)
- 常规方法: 总读数归一化(Total Count)、中位数/分位数归一化。
- CRISPR特定优化: 基于内参 sgRNA 或非靶向对照(Non-targeting Control)的归一化。
- 极端样本处理: 针对高细胞致死率或强选择压力样本,选择更稳健的算法,并通过分布折线图/直方图验证归一化效果。
- 核心思路: 确保不同样本之间的数据可比,即使实验条件复杂或有异常数据,也能得到可靠结果。
4. 覆盖度与测序深度分析(Coverage & Depth)
- 指标定义: sgRNA 覆盖度指文库的完整性;读数深度指测序数据的丰度。
- 实验标准: 进行sgRNA 分布统计、sgRNA比对文库匹配率可视化,可以有效预警低质量样本,也可以计算GINI指数用以评估文库均一性。
- 核心思路: 文库设计合理、测序充分,才能保证筛选结果科学可靠。
总结
在选择 CRISPR 文库分析工具时,建议从 灵敏度、特异性、假发现率控制、归一化稳健性、文库覆盖度与测序深度 这几个维度综合考量。这样既能保证数据质量,又能降低下游验证成本,为科研决策提供坚实依据。
五、基于实验条件的 CRISPR 文库分析工具选型策略
不同实验设计会带来不同的数据分析挑战,因此在选择分析工具或方法时,应根据实验条件采取差异化策略,以确保结果可靠、可解释。以下是常见实验场景及对应建:
1. 小样本/低重复数(如 n=2)
- 潜在风险: 样本量过少会导致统计效力不足,方差估计不稳定,容易漏检或误检候选基因。
- 分析策略: 采用借用整体信息(Information Borrowing)的统计方法(如经验贝叶斯估计、负二项回归)。利用 iScreenAnlys™文库分析平台的通路富集分析模块,增强单基因结果的生物学解释力。
- 核心思路: 用统计方法“放大”信息量,同时结合通路分析增加可靠性。
2. 大样本/复杂设计(多时间点、多剂量)
- 潜在风险: 批次效应、未建模协变量等因素可能干扰真实生物信号
- 分析策略: 利用 DESeq2 / edgeR 的广义线性模型(Generalized Linear Model, GLM)处理复杂设计。配置对比矩阵与协变量,并通过 PCA/聚类分析诊断并校正批次效应。
- 核心思路: 用成熟统计建模方法分离真实信号和技术噪音,确保复杂设计下的分析可靠性。
3. 低测序深度/文库覆盖度不足
- 潜在风险: 弱效应基因容易漏检,统计结论波动大。
- 分析策略: 严格执行 QC 流程,确认样本可用性。避免使用对低计数高度敏感的算法,聚焦于高效应量基因及通路层面的信号。
- 核心思路: 保证分析的可靠性,即使文库或测序不理想,也能挖掘核心信息。
4. 资源受限(预算/专业人员缺乏)
- 潜在风险: 难以维护复杂的生信流程,分析效率低、易出错。
- 分析策略: 选择高自动化、界面友好的集成平台,如 iScreenAnlys™文库分析平台,该平台对 MAGeCK、DESeq2 等工具封装标准化操作流程(SOP),降低学习和维护成本。
总结
选择分析方法不仅看工具功能,还要结合实验条件:样本量、实验设计复杂性、测序深度和资源状况。根据不同场景采取差异化策略,可以显著提高 CRISPR 文库筛选数据的可靠性和可解释性。在这一过程中,源井生物 iScreenAnlys™ 文库分析平台凭借一站式端到端流程、CRISPR 专用统计模型、可视化交互界面以及高效团队协作能力,成为科研人员进行 CRISPR 文库分析的理想选择,让研究更高效、更稳健、更可控。
六、 常见分析偏差与规避策略
在 CRISPR 文库数据分析中,科研人员容易陷入一些常见误区。了解这些偏差并采取相应措施,可以显著提高分析结果的可靠性与可解释性。
1. 单维度指标依赖
- 误区: 仅关注 p 值 或 FDR,忽略信号强度和一致性
- 规避: 应综合考量对数变化倍数(logFC)、sgRNA 效应的一致性及通路富集结果。利用多维可视化图表进行交叉验证。
- 核心思路: 不仅看“显著性”,还要看“生物学意义”。
2. 忽视前置质控
- 误区: 直接跳过质控(QC)进行差异分析。
- 规避: 必须优先检查 sgRNA 覆盖度/mapped率、GINI指数及样本相关性。QC 是分析流程的必经环节。
- 核心思路: 数据质量是分析可靠性的基础。
3. 分析流程的不一致性
- 误区: 在同一研究中随意切换不同分析方法。
- 规避: 建立并遵循标准化的分析模板,确保项目内部及项目间结果的可比性。
- 核心思路: 流程统一,结果才可靠。
4. “黑箱”工具的盲目使用
- 误区: 不理解算法假设,直接使用工具输出结果。
- 规避: 参考分析工具提供的说明文档,或者寻求社区支持,理解模型的适用范围与局限性。
- 核心思路: 理解原理,才能科学使用工具
总结
CRISPR 文库分析不仅是数据处理,更是科学判断与方法选择的结合。通过关注多维指标、严格 QC、标准化流程和合理使用工具,可以最大限度保证分析结果的可信度和可解释性。
七、 结论与实践探讨
1. 综合结论
经典工具(如 MAGeCK、DESeq2/edgeR)为 CRISPR Screen 数据分析奠定了坚实的统计学基础,方法成熟、可靠,已被科研界广泛验证。而 iScreenAnlys™ 文库分析平台 的核心优势并非重新发明轮子,而是通过 工程化集成 将这些方法发挥得更彻底。平台将严谨的统计内核 封装进统一且可视化的分析工作流,实现从 数据质控 → 模型分析 → 结果解释 的端到端覆盖。对于绝大多数研究场景,iScreenAnlys™ 以标准化方式调用并增强经典算法,使分析流程更稳定、参数更透明、结果更可复现。
在保证学术严谨性的前提下,iScreenAnlys™ 文库分析平台显著提升了科研效率与多项目管理能力,让研究者能够更加专注于科学问题本身,而非被繁琐的工具链困扰。换句话说,iScreenAnlys™ 并非替代经典工具,而是让已有方法更高效、更易用、更可靠。
2. 关键实践问答(FAQ)
-
Q1:如果已经熟练使用 MAGeCK,为什么还要迁移到 iScreenAnlys™?
A: iScreenAnlys™文库分析平台并非排他性的替代品,而是对 MAGeCK 等工具的智能化封装。它在保留核心统计方法不变的前提下,提供了更为完善的质控体系、交互式可视化及项目管理功能,实质上是对现有流程的效能升级。 -
Q2:如何处理小样本或低覆盖度数据?
A: 此类数据仍可分析,但需保持谨慎。iScreenAnlys™文库分析平台的 QC 模块会能揭示深度与覆盖度缺陷,辅助研究者客观评估数据的局限性。 -
Q3:平台是否适合非生物信息背景的实验人员?
A: 完全适合。iScreenAnlys™文库分析平台的设计初衷即是降低技术门槛,使实验人员能通过图形界面执行符合行业标准的分析流程。
总结
iScreenAnlys™ 文库分析平台是对经典 CRISPR Screen 分析方法的智能升级:高效、可靠、可复现,同时降低技术门槛。无论是小规模实验还是复杂项目,都能帮助科研人员快速、准确地从数据中挖掘关键生物学信息。
立即预约免费试用 iScreenAnlys™ 文库分析平台。体验真正的一站式 CRISPR 文库分析流程:从原始数据导入、质控、归一化,到差异分析、可视化与结果解读,全流程高效完成,让科研更专注于科学,而非繁琐操作。










