当前位置:首页 > 技术服务 > CRISPR-U™ 基因编辑细胞 >



独家创新技术,
基因编辑效率提升10倍;

超过100种细胞
基因编辑成功案例;

轻松实现基因敲除
基因点突变和基因敲入;

承诺项目失败全额退款,
成功保障安全无忧;
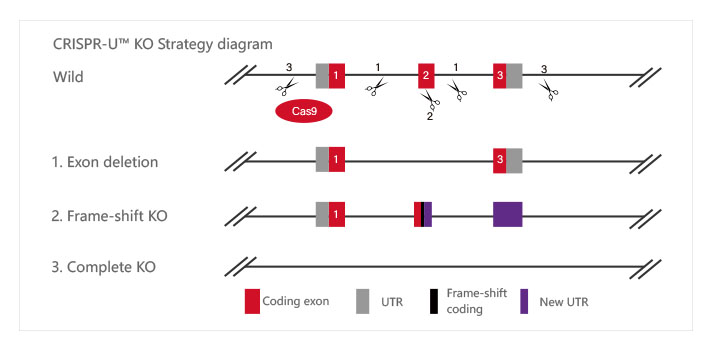
· 常见基因敲除细胞系类型
| 类 别 | |
|---|---|
| 敲除片段大小 | 小片段敲除;移码敲除;片段敲除(见下图) |
| 转染方法 | 病毒法;化学转染法;电转染法 |
| 细胞类型 | 肿瘤细胞;其他细胞系;IPS/ES等 |
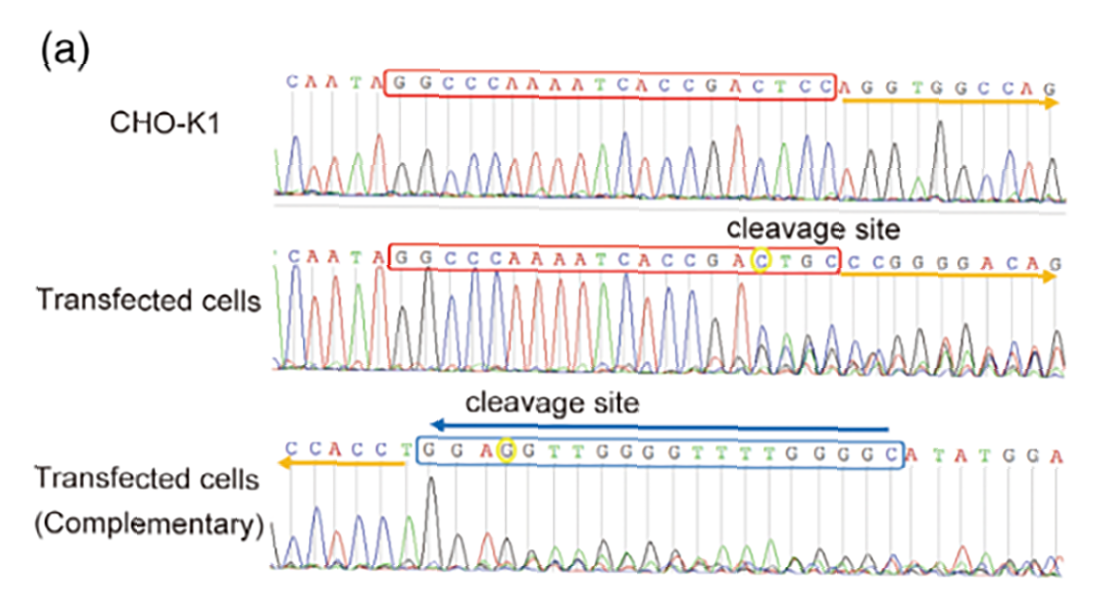
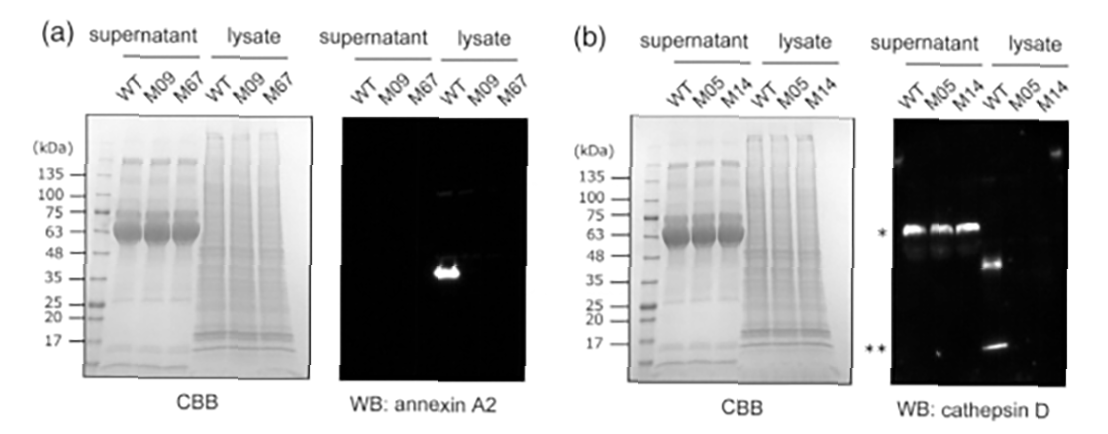
(a)sgRNA打靶Anxa2基因的4号外显子。
对序列进行双向分析。
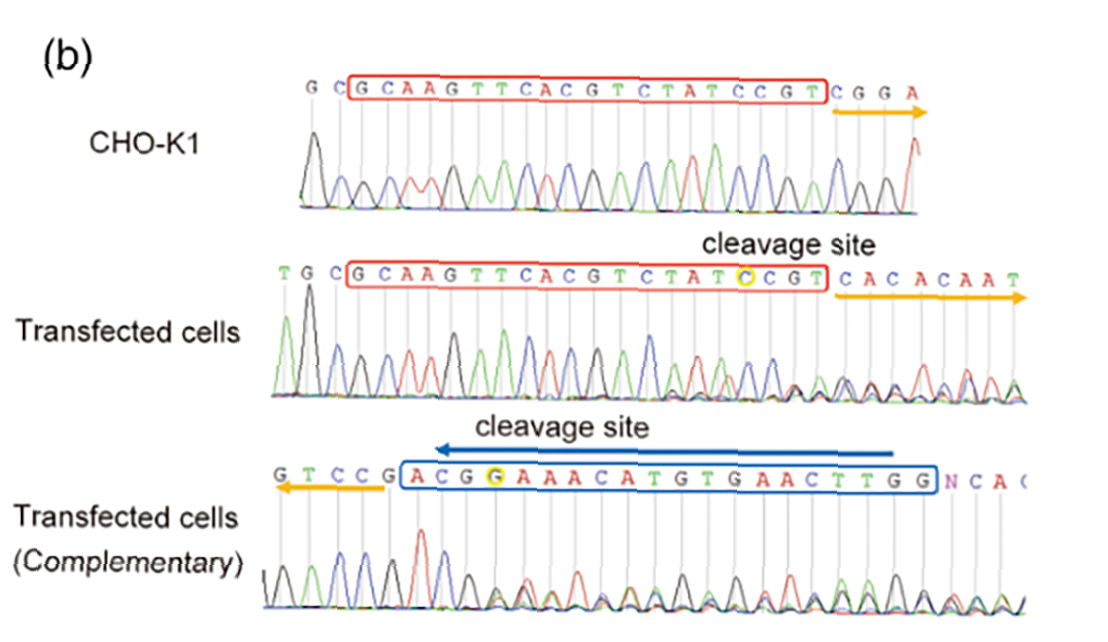
(b) sgRNA打靶Ctsd基因2号外显子。
对序列进行双向分析。